Nyt værktøjssæt gør simuleringer af molekylær dynamik mere tilgængelige
Molekylær dynamik (MD) simuleringer er blevet et stærkt værktøj inden for de stadigt voksende områder inden for molekylærbiologi og lægemiddeludvikling. Mens der findes mange MD-simuleringsteknikker, er parallel kaskadeselektion MD (PaCS-MD) en særlig nyttig, når man studerer foldningen (eller "konformationen") af proteiner eller interaktionerne mellem proteiner og ligander.
Kernen i denne metode består i at køre flere MD-simuleringer parallelt og derved udforske forskellige mulige konformationer samtidigt. Ved hjælp af omhyggeligt udformede udvælgelseskriterier kan lovende konformationer detekteres automatisk i "temporale snapshots" og undersøges nærmere. Denne strategi fremskynder i høj grad opdagelsen af vigtige molekylære interaktioner og dynamiske processer, der hjælper videnskabsmænd med at forstå proteinfunktionelle bevægelser.
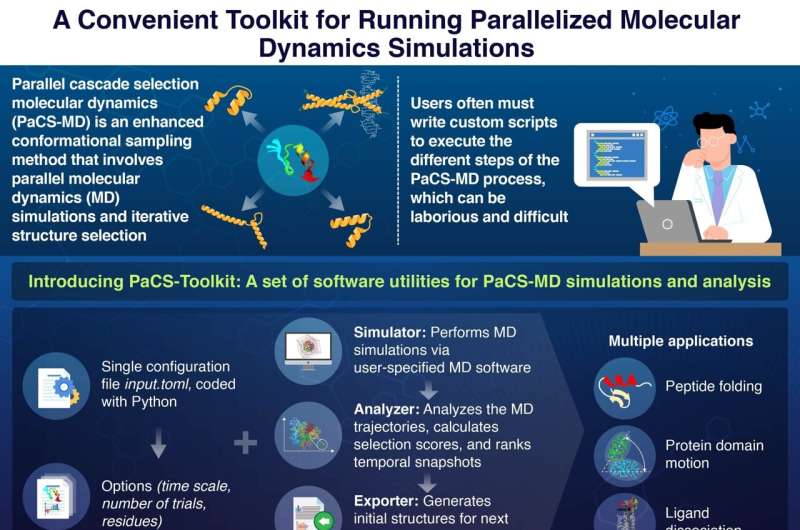
En af forhindringerne ved PaCS-MD er imidlertid, at brugerne skal skrive brugerdefinerede scripts for at udføre deres ønskede MD-simuleringer. I disse scripts skal de specificere startbetingelserne og målfunktionerne, vælge MD-software til brug, implementere en snapshot-rangeringsprocedure og forberede de indledende strukturer til den næste simuleringscyklus. Denne proces, som kan være ret kompleks og udsat for fejl, skaber en betydelig adgangsbarriere for forskere, der er interesseret i at bruge PaCS-MD.
Heldigvis har et team af forskere fra School of Life Science and Technology ved Tokyo Institute of Technology, Japan, for nylig sat sig for at løse dette problem. I deres seneste undersøgelse, som blev offentliggjort i The Journal of Physical Chemistry B og ledet af professor Akio Kitao udviklede holdet en softwarepakke kaldet PaCS-Toolkit for at gøre PaCS-MD mere tilgængelig og praktisk at bruge.
En bemærkelsesværdig fordel ved PaCS-Toolkit er, at hele simuleringsprocessen er sat op via en enkelt konfigurationsfil. I denne fil specificerer brugerne vigtige parametre for simuleringerne, herunder typen af PaCS-MD, antallet af MD-simuleringer, der skal køres parallelt, og de proteinrester eller atomer, der skal holdes styr på som udvælgelseskriterier for parallelle forgreninger.
PaCS-Toolkit tager denne konfigurationsfil sammen med standard MD-inputfiler og kører PaCS-MD-simuleringer i henhold til den specificerede MD-software. Værd at bemærke, at da pakken er åben software skrevet i Python – et populært programmeringssprog – kan brugere hjælpe med at forbedre PaCS-Toolkit og udvide dets funktionaliteter.
"Vores værktøjssæt bevarer fleksibiliteten, så nye funktioner, biblioteker og MD-software kan tilføjes ved at introducere ansvarlige klasser i Python. Brugere, der kan programmere på dette sprog, bør være i stand til at ændre koden for PaCS-Toolkit og implementere nye metoder efter behov, " siger prof. Kitao.
En anden afgørende fordel ved PaCS-Toolkit ligger i dets optimering og kompatibilitet med forskellige computermiljøer, det være sig en række supercomputere med en beskedoverførselsgrænseflade (MPI), servere udstyret med flere grafikkort (GPU'er) eller personlige computere såsom bærbare computere.
"PaCS-Toolkit inkorporerer i høj grad parallelisering ved hjælp af MPI, GPU og multiprocessing-pakken i Python, hvilket muliggør optimering af beregningstiden afhængigt af de tilgængelige beregningsressourcer," forklarer Prof. Kitao.
For at vise potentialet i deres værktøjskasse gennemførte forskerne PaCS-MD-simuleringer skræddersyet til tre forskellige applikationer. Disse omfattede foldningen af mini-proteinet chignolin, bevægelsen af proteindomæner i et SARS-CoV-2-enzym og liganddissociation fra en vigtig adenosinreceptor.
"Tilsammen indikerer vores resultater, at PaCS-Toolkit let kan bruges til at simulere en bred vifte af dynamikker for forskellige typer af molekylære systemer," konkluderer Prof. Kitao.
Dette værktøjssæt kan hjælpe med at frigøre det sande potentiale af PaCS-MD-simuleringer på forskellige områder, hvilket gør det muligt for interesserede forskere at kaste lys over komplekse molekylære processer og fremskynde opdagelse af lægemidler.
Flere oplysninger: Shinji Ikizawa et al., PaCS-Toolkit:Optimized Software Utilities for Parallel Cascade Selection Molecular Dynamics (PaCS-MD) Simulations and Subsequent Analyses, The Journal of Physical Chemistry B (2024). DOI:10.1021/acs.jpcb.4c01271
Journaloplysninger: Journal of Physical Chemistry B
Leveret af Tokyo Institute of Technology
 Varme artikler
Varme artikler
-
 Forskere udvikler en diagnostisk test, som har til formål at påvise antibiotikaresistens på mindr…Kredit:Medicinsk Forskningsråd Forskere udvikler en lav pris, hurtig diagnostisk sensortest, som har til formål at vise bakteriers modtagelighed for antibiotika inden for 45 minutter. NHS laborat
Forskere udvikler en diagnostisk test, som har til formål at påvise antibiotikaresistens på mindr…Kredit:Medicinsk Forskningsråd Forskere udvikler en lav pris, hurtig diagnostisk sensortest, som har til formål at vise bakteriers modtagelighed for antibiotika inden for 45 minutter. NHS laborat -
 Ny eksfolieringsteknik genvinder biomaterialers piezoelektricitetZhang Zhuomin, et medlem af Dr. Yang Zhengbaos forskerhold, demonstrerer råmaterialet i tyndtarmens submucosa fra får. Kredit:City University of Hong Kong Piezoelektriske materialer er anvendelige
Ny eksfolieringsteknik genvinder biomaterialers piezoelektricitetZhang Zhuomin, et medlem af Dr. Yang Zhengbaos forskerhold, demonstrerer råmaterialet i tyndtarmens submucosa fra får. Kredit:City University of Hong Kong Piezoelektriske materialer er anvendelige -
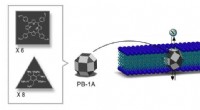 Syntetiske nanokanaler til jodidtransport3-D struktur af den syntetiske iodid-transporter PB-1A udviklet af IBS forskere. PB-1A-design følger formen af 26-sidet polyeder lavet af trekanter og firkanter (mørkegrå) med 12 flader efterladt to
Syntetiske nanokanaler til jodidtransport3-D struktur af den syntetiske iodid-transporter PB-1A udviklet af IBS forskere. PB-1A-design følger formen af 26-sidet polyeder lavet af trekanter og firkanter (mørkegrå) med 12 flader efterladt to -
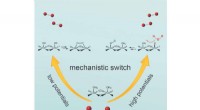 Potentialafhængig afbryder hjælper med vandopdeling ved hjælp af koboltoxid-katalysatorerEn potentialafhængig afbryder hjælper med vandspaltning ved hjælp af cobaltoxid-katalysatorer, Boston College og Yale University-forskere rapporterer i tidsskriftet Chem. Lille elektrisk ladning drive
Potentialafhængig afbryder hjælper med vandopdeling ved hjælp af koboltoxid-katalysatorerEn potentialafhængig afbryder hjælper med vandspaltning ved hjælp af cobaltoxid-katalysatorer, Boston College og Yale University-forskere rapporterer i tidsskriftet Chem. Lille elektrisk ladning drive
- Forskere udvikler grønnere og mere effektiv metode til fremstilling af næste generations antibioti…
- Vridning af grafen til spiraler - forskere syntetiserer spiralformet nanograf
- Zinkoxid nanotråde:Ny løsning til billigere, renere produktion af elektroniske komponenter
- MagicMark:En markeringsmenu ved hjælp af 2-D-retning og 3D-dybdeinformation
- En ny praktisk test for funktionen af HDL, bæreren af det gode kolesterol
- Sådan beregnes terningssandsynligheder


