Hvordan har du prøvet at slukke og tænde den igen? arbejder for kemi, ikke kun computere
En ny undersøgelse fra Tel Aviv University har opdaget, at en kendt praksis inden for informationsteknologi også kan anvendes til kemi. Forskere fandt ud af, at for at forbedre prøvetagningen i kemiske simuleringer, skal du blot stoppe og genstarte.
Forskningen blev ledet af ph.d. studerende Ofir Blumer, i samarbejde med professor Shlomi Reuveni og Dr. Barak Hirshberg fra Sackler School of Chemistry ved Tel Aviv Universitet. Undersøgelsen blev offentliggjort i Nature Communications .
Forskerne forklarer, at simuleringer af molekylær dynamik er som et virtuelt mikroskop. De sporer bevægelsen af alle atomer i kemiske, fysiske og biologiske systemer, såsom proteiner, væsker og krystaller. De giver indsigt i forskellige processer og har forskellige teknologiske anvendelser, herunder lægemiddeldesign.
Disse simuleringer er dog begrænset til processer, der er langsommere end en milliontedel af et sekund og kan således ikke beskrive langsommere processer såsom proteinfoldning og krystalkernedannelse. Denne begrænsning, kendt som tidsskalaproblemet, er en stor udfordring i marken.
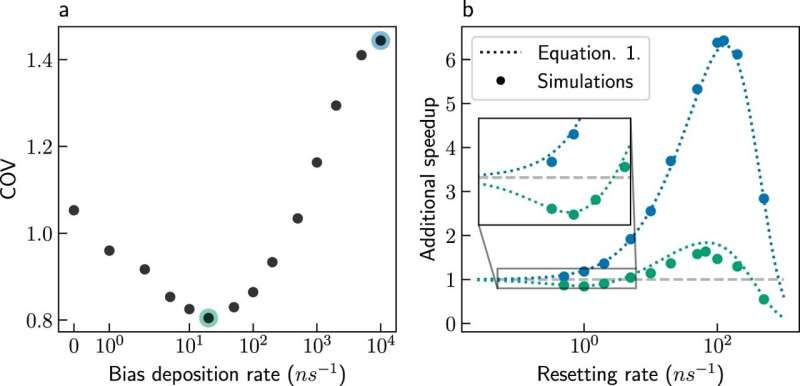
Ph.D. studerende Ofir Blumer siger:"I vores nye undersøgelse viser vi, at tidsskalaproblemet kan overvindes ved stokastisk nulstilling af simuleringerne. Det virker kontraintuitivt ved første øjekast - hvordan kan simuleringerne ende hurtigere, når de genstartes? Alligevel viser det sig, at reaktionen tider varierer betydeligt mellem simuleringer. I nogle simuleringer forekommer reaktioner hurtigt, men andre simuleringer forsvinder i mellemtilstande i lange perioder. Nulstilling forhindrer simuleringer i at sidde fast i sådanne mellemprodukter og forkorter den gennemsnitlige simuleringstid."
Forskerne kombinerede også stokastisk nulstilling med Metadynamics, en populær metode til at fremskynde simuleringer af langsomme kemiske processer. Kombinationen tillader større acceleration end hver metode separat. Desuden er Metadynamics afhængig af forudgående viden:reaktionskoordinaterne skal være kendt for at fremskynde simuleringen.
Kombinationen af Metadynamics med nulstilling reducerer afhængigheden af forudgående viden betydeligt, hvilket sparer tid for udøvere af metoden. Endelig viste forskerne, at kombinationen giver mere præcise forudsigelser af hastigheden af langsomme processer. Den kombinerede metode blev brugt til at forbedre simuleringer af et proteinfoldning i vand med succes, og det forventes at blive anvendt på flere systemer i fremtiden.
Flere oplysninger: Ofir Blumer et al., Kombination af stokastisk nulstilling med metadynamics for at fremskynde simuleringer af molekylær dynamik, Nature Communications (2024). DOI:10.1038/s41467-023-44528-w
Journaloplysninger: Nature Communications
Leveret af Tel-Aviv University
 Varme artikler
Varme artikler
-
 Grøn kemi og biobrændstof:Mekanismen for et centralt fotoenzym dekrypteretKunstnerindtryk af den enzymatiske katalyse foreslået i mekanisme af fedtsyrefotodecarboxylase. Kredit:Damien Sorigué Funktionen af enzymet FAP, nyttig til produktion af biobrændstoffer og til g
Grøn kemi og biobrændstof:Mekanismen for et centralt fotoenzym dekrypteretKunstnerindtryk af den enzymatiske katalyse foreslået i mekanisme af fedtsyrefotodecarboxylase. Kredit:Damien Sorigué Funktionen af enzymet FAP, nyttig til produktion af biobrændstoffer og til g -
 Bygge vægge, der vil gøre sommerens hedebølger mere tålelige(a) PCM-vægtværsnit (b) temperaturmålingsposition. Kredit:Korea Institute of Science and Technology (KIST) Med sommervarmen, der bliver mere og mere uudholdelig og langvarig på grund af klimaændri
Bygge vægge, der vil gøre sommerens hedebølger mere tålelige(a) PCM-vægtværsnit (b) temperaturmålingsposition. Kredit:Korea Institute of Science and Technology (KIST) Med sommervarmen, der bliver mere og mere uudholdelig og langvarig på grund af klimaændri -
 Video:Hvordan kemi gør fjernelse af kuldioxid muligKredit:The American Chemical Society I løbet af de sidste par hundrede år, mængden af kuldioxid i vores atmosfære er steget i vejret. Hvis vi ikke fjerner i det mindste noget af det, der kommer
Video:Hvordan kemi gør fjernelse af kuldioxid muligKredit:The American Chemical Society I løbet af de sidste par hundrede år, mængden af kuldioxid i vores atmosfære er steget i vejret. Hvis vi ikke fjerner i det mindste noget af det, der kommer -
 Ny strategi udviklet til redox divergerende kobling af ketoner med terpenerPd-katalyseret redox divergerende kobling af ketoner med terpenoler. Kredit:ZHAO Chaoyang Hydrogenering og dehydrogenering er to vigtige kunstige metoder til at ændre redoxtilstanden af forbinde
Ny strategi udviklet til redox divergerende kobling af ketoner med terpenerPd-katalyseret redox divergerende kobling af ketoner med terpenoler. Kredit:ZHAO Chaoyang Hydrogenering og dehydrogenering er to vigtige kunstige metoder til at ændre redoxtilstanden af forbinde
- Sådan løses matematiske problemer ved hjælp af en Flowchart
- Vindmøllepark ydeevne-forudsigelse og optimering spotlight
- Sådan forbereder du dit hjem til sommerens hedebølger
- Ny bog udforsker, hvordan pesten formede kristendommen i Amerika
- Farlige kemikalier er uregulerede i rutinemæssige olie- og gasoperationer
- Kunne fri energi kun være en skylning væk?


