Forskere bygger en enzym-opdagende AI
Mens E. coli er en af de mest undersøgte organismer, er funktionen af 30 % af proteinerne, der udgør E. coli, endnu ikke blevet klart afsløret. Til dette blev en kunstig intelligens brugt til at opdage 464 typer enzymer fra proteinerne, der var ukendte, og forskerne fortsatte med at verificere forudsigelserne om tre typer proteiner, der med succes blev identificeret gennem in vitro enzymassay.
Et fælles forskerhold, herunder Gi Bae Kim, Ji Yeon Kim, Dr. Jong An Lee og den fremtrædende professor Sang Yup Lee fra Institut for Kemi- og Biomolekylær Teknik ved KAIST, og Dr. Charles J. Norsigian og professor Bernhard O. Palsson fra Institut for Bioteknik på UCSD, har udviklet DeepECtransformer, en kunstig intelligens, der kan forudsige enzymfunktionerne ud fra proteinsekvensen. Derudover har holdet etableret et forudsigelsessystem ved at bruge AI til hurtigt og præcist at identificere enzymfunktionen.
Holdets arbejde er beskrevet i papiret med titlen "Funktionel annotering af enzymkodende gener ved hjælp af dyb læring med transformerlag." Avisen blev offentliggjort den 14. november i Nature Communications .
Enzymer er proteiner, der katalyserer biologiske reaktioner, og identifikation af funktionen af hvert enzym er afgørende for at forstå de forskellige kemiske reaktioner, der findes i levende organismer, og disse organismers metaboliske egenskaber.
Enzyme Commission (EC) nummer er et enzymfunktionsklassifikationssystem designet af International Union of Biochemistry and Molecular Biology, og for at forstå forskellige organismers metaboliske egenskaber er det nødvendigt at udvikle en teknologi, der hurtigt kan analysere enzymer og EC-numre af enzymerne til stede i genomet.
Forskellige metoder baseret på dyb læring er blevet udviklet til at analysere funktionerne i biologiske sekvenser, herunder forudsigelse af proteinfunktioner, men de fleste af dem har et problem med en sort boks, hvor inferensprocessen af AI ikke kan fortolkes.
Forskellige forudsigelsessystemer, der anvender AI til forudsigelse af enzymfunktion, er også blevet rapporteret, men de løser ikke dette sorte boks-problem eller kan ikke fortolke ræsonnementsprocessen på et finkornet niveau (f.eks. niveauet af aminosyrerester i enzymsekvensen ).
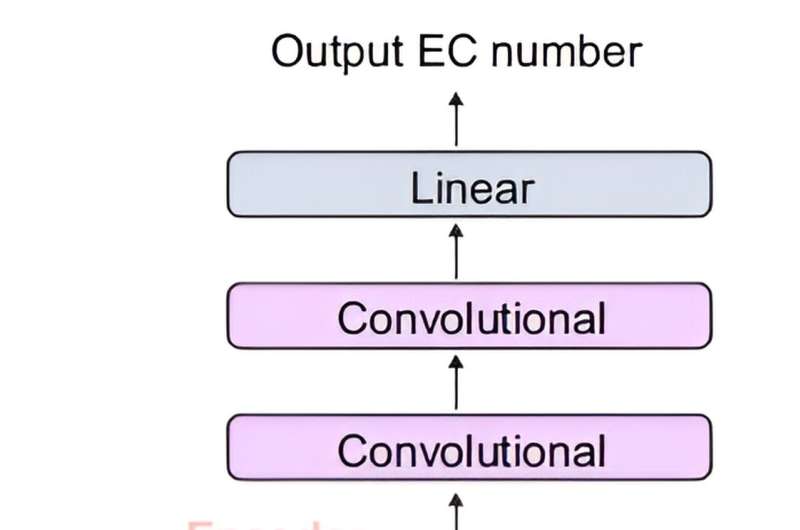
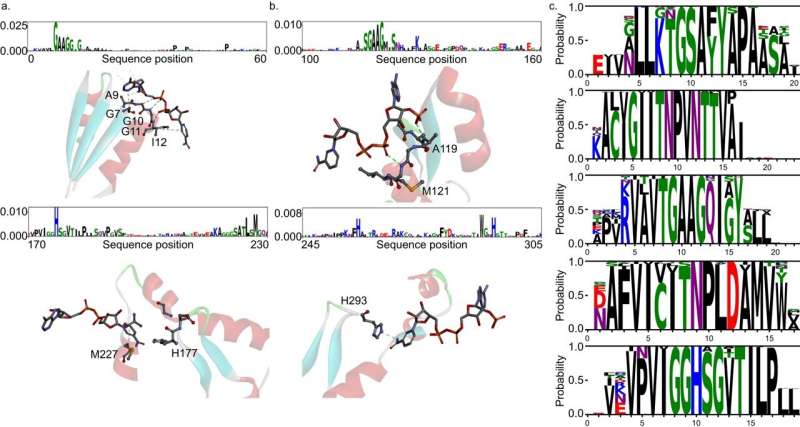
Det fælles team udviklede DeepECtransformer, en kunstig intelligens, der anvender deep learning og et proteinhomologianalysemodul til at forudsige enzymfunktionen af en given proteinsekvens.
For bedre at forstå funktionerne i proteinsekvenser blev transformatorarkitekturen, som almindeligvis bruges i naturlig sprogbehandling, desuden brugt til at udtrække vigtige funktioner om enzymfunktioner i sammenhæng med hele proteinsekvensen, hvilket gjorde det muligt for teamet at præcist forudsige EC nummeret på enzymet. Den udviklede DeepECtransformer kan forudsige i alt 5360 EC-numre.
Det fælles team analyserede yderligere transformatorarkitekturen for at forstå inferensprocessen i DeepECtransformer og fandt ud af, at AI i inferensprocessen anvender information om katalytiske aktive steder og/eller cofaktorbindingssteder, som er vigtige for enzymfunktionen. Ved at analysere den sorte boks af DeepECtransformer blev det bekræftet, at AI var i stand til at identificere de funktioner, der er vigtige for enzymfunktionen alene under indlæringsprocessen.
"Ved at bruge det forudsigelsessystem, vi udviklede, var vi i stand til at forudsige funktionerne af enzymer, der endnu ikke var blevet identificeret, og verificere dem eksperimentelt," sagde Gi Bae Kim, den første forfatter af papiret.
"Ved at bruge DeepECtransformer til at identificere hidtil ukendte enzymer i levende organismer, vil vi være i stand til mere præcist at analysere forskellige facetter involveret i organismers metaboliske processer, såsom de enzymer, der er nødvendige for at biosyntetisere forskellige nyttige forbindelser eller de enzymer, der er nødvendige for at bionedbryde plast." tilføjede han.
"DeepECtransformer, som hurtigt og præcist forudsiger enzymfunktioner, er en nøgleteknologi inden for funktionel genomik, som gør os i stand til at analysere funktionen af hele enzymer på systemniveau," sagde professor Sang Yup Lee.
Han tilføjede:"Vi vil være i stand til at bruge det til at udvikle miljøvenlige mikrobielle fabrikker baseret på omfattende metaboliske modeller i genomskala, hvilket potentielt minimerer manglende information om metabolisme."
Flere oplysninger: Gi Bae Kim et al., Funktionel annotering af enzymkodende gener ved hjælp af deep learning med transformerlag, Nature Communications (2023). DOI:10.1038/s41467-023-43216-z
Journaloplysninger: Nature Communications
Leveret af Korea Advanced Institute of Science and Technology (KAIST)
 Varme artikler
Varme artikler
-
 Farveskiftende gnisterKredit:Clausthal University of Technology Gnister er et fascinerende fænomen, der er kendt fra lejrbål, flintsten og elektriske stjernekast og andre pyrotekniske artikler. Når man ser nærmere på g
Farveskiftende gnisterKredit:Clausthal University of Technology Gnister er et fascinerende fænomen, der er kendt fra lejrbål, flintsten og elektriske stjernekast og andre pyrotekniske artikler. Når man ser nærmere på g -
 Kemikere foreslår en ny måde at syntetisere steroidanaloger påIntramolekylær Diels-Alder reaktion diskuteret i forskningen. Kredit:Fedor Zubkov Forskere fra RUDN University og Ivan Franko National University of Lviv har fundet en måde at fremstille aromatisk
Kemikere foreslår en ny måde at syntetisere steroidanaloger påIntramolekylær Diels-Alder reaktion diskuteret i forskningen. Kredit:Fedor Zubkov Forskere fra RUDN University og Ivan Franko National University of Lviv har fundet en måde at fremstille aromatisk -
 For at opsnuse kriminalitet og forsvundne personer, videnskaben støtter bloddetektionshundeHunde kan pålideligt opsnuse menneskeblod, selv efter to års miljøforringelse. Kredit:Jason Korbol/shutterstock Det er svært at overveje tragedien ved at miste en elsket en og aldrig vide, hvad de
For at opsnuse kriminalitet og forsvundne personer, videnskaben støtter bloddetektionshundeHunde kan pålideligt opsnuse menneskeblod, selv efter to års miljøforringelse. Kredit:Jason Korbol/shutterstock Det er svært at overveje tragedien ved at miste en elsket en og aldrig vide, hvad de -
 Selektiv opløsning af elementært guld fra multimetalkilder i organiske opløsningerKredit:Angewandte Chemie Urban minedrift, genanvendelse af ædle metaller fra elektroniske gadgets, bliver stadig vigtigere, selvom processer, der er både effektive og miljøvenlige, stadig er få. E
Selektiv opløsning af elementært guld fra multimetalkilder i organiske opløsningerKredit:Angewandte Chemie Urban minedrift, genanvendelse af ædle metaller fra elektroniske gadgets, bliver stadig vigtigere, selvom processer, der er både effektive og miljøvenlige, stadig er få. E
- Forskning viser, hvor meget administrerende direktører betyder noget for virksomhedens præstatione…
- Forskere søger atomistisk indsigt i ferroelektriske materialer
- Forsker afslører, hvordan stereotyper om brillans former kvinders beslutninger om at studere psykol…
- Forskere finder en måde at skabe lang levetid, hurtigopladelige batterier
- Græseksperter forklarer, hvorfor mulching af blade er en bedre løsning end bortskaffelse
- Forskere genbesøger det kolde tilfælde af kold fusion


