Nanopore-teknik afkoder hurtigt lange DNA-strenge
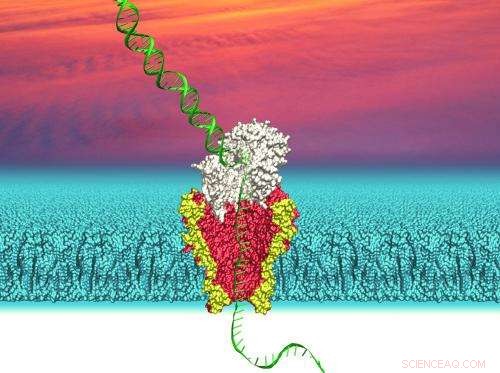
Illustration af en nanopore afledt af en genetisk modificeret bakteriel membrankanal, der bruges til at sekventere DNA. Kredit:Ian Derrington
(Phys.org) —En lavpristeknologi kan gøre det muligt at læse lange DNA-sekvenser langt hurtigere end nuværende teknikker.
Forskningen fremmer en teknologi, kaldet nanopore DNA-sekventering. Hvis det perfektioneres, kunne det en dag bruges til at skabe håndholdte enheder, der hurtigt kan identificere DNA-sekvenser fra vævsprøver og miljøet, sagde forskere fra University of Washington, der udviklede og testede tilgangen.
"En grund til, at folk er så begejstrede for nanopore DNA-sekventering, er, at teknologien muligvis kan bruges til at skabe 'tricorder'-lignende enheder til at opdage patogener eller diagnosticere genetiske lidelser hurtigt og på stedet, " sagde Andrew Laszlo, hovedforfatter og kandidatstuderende i Jen Gundlachs laboratorium, en UW professor i fysik, der ledede projektet.
Papiret "Decoding long nanopore sequencing reads of natural DNA" beskriver den nye teknik. Det fremgår 25. juni i den avancerede onlineudgave af tidsskriftet Natur bioteknologi .
De fleste af de nuværende gensekventeringsteknologier kræver arbejde med korte DNA-stykker, typisk 50 til 100 nukleotider lange. Disse skal behandles af store sequencere i et laboratorium. Den besværlige proces kan tage dage til uger at gennemføre.
Nanopore teknologi udnytter de små, tunnellignende strukturer fundet i bakterielle membraner. I naturen, sådanne porer tillader bakterier at kontrollere strømmen af næringsstoffer over deres membraner.
UW-forsker brugte nanopore Mycobacterium smegmatis porin A (MspA). Denne bakterielle pore er blevet genetisk ændret, så den smalleste del af kanalen har en diameter på omkring en nanometer, eller 1 milliardtedel af en meter. Dette er stort nok til, at en enkelt DNA-streng kan passere igennem. Den modificerede nanopore indsættes derefter i en membran, der adskiller to saltopløsninger for at skabe en kanal, der forbinder de to opløsninger.
For at læse en sekvens af DNA med dette system, en lille spænding påføres over membranen for at få ionerne i saltopløsningen til at strømme gennem nanoporen. Ionstrømmen skaber en målbar strøm. Hvis en DNA-streng tilsættes opløsningen på den ene side af membranen og derefter kommer ind i en pore, de omfangsrige DNA-molekyler vil hæmme strømmen af den meget mindre ion og derved ændre strømmen. Hvor meget strømmen ændrer sig afhænger af hvilke nukleotider – de enkelte molekyler adenin, guanin, cytosin og thymin, der udgør DNA-kæden - er inde i poren. Detektering af ændringer i strøm kan afsløre, hvilke nukleotider der passerer gennem nanoporens kanal på et givet tidspunkt.
Siden teknikken først blev foreslået i 1990'erne, forskere håbede, at nanopore DNA-sekventering ville tilbyde en billig, hurtigt alternativ til nuværende gensekventering. Men deres forsøg er blevet frustreret af flere udfordringer. Det er svært at identificere hvert nukleotid en for en, når de passerer gennem nanoporen. I stedet, forskere skal arbejde med ændringer i strøm forbundet med fire nukleotider ad gangen. Ud over, nogle nukleotider kan mangle eller læse mere end én gang. Følgelig, nuværende nanopore-sekventeringsteknologi giver en upræcis udlæsning af en DNA-sekvens.
UW-forskerne beskriver, hvordan de omgik disse problemer. Forskerne identificerede først de elektroniske signaturer af alle mulige nukleotidkombinationer med de fire nukleotider, der udgør DNA - i alt 256 kombinationer (4 x 4 x 4 x 4).
De skabte derefter computeralgoritmer til at matche de aktuelle ændringer, der genereres, når et segment af DNA passerer gennem poren med aktuelle ændringer, der forventes fra DNA-sekvenser af kendte gener og genomer, der er lagret i en computerdatabase. Et match ville vise, at sekvensen af DNA'et, der passerede gennem poren, var identisk med eller tæt på DNA-sekvensen, der er lagret i databasen. Hele processen ville tage minutter til et par timer, i stedet for uger.
For at teste denne tilgang, forskerne brugte deres nanopore-system til at læse sekvensen af bakteriofag Phi X 174, en virus, der inficerer bakterier, og som almindeligvis bruges til at evaluere nye genomsekventeringsteknologier. De fandt ud af, at metoden pålideligt læste bakteriofagens DNA-sekvenser og kunne læse sekvenser så længe som 4, 500 nukleotider.
"Dette er første gang nogen har vist, at nanoporer kan bruges til at generere fortolkbare signaturer, der svarer til meget lange DNA-sekvenser fra den virkelige verdens genomer, " sagde medforfatter Jay Shendure, en UW lektor i genomvidenskab, hvis laboratorium udvikler anvendelser af genomsekventeringsteknologier. "Det er et stort skridt fremad."
Fordi teknikken er afhængig af at matche aflæsninger til databaser med tidligere sekventerede gener og genomer, det kan endnu ikke bruges til at sekventere et nyopdaget gen eller genom, sagde forskerne, men med nogle justeringer, tilføjede de, det burde være muligt at forbedre ydeevnen på dette område. For at fremskynde forskningen i denne nye teknologi, forskerne laver deres metoder, data og computeralgoritmer fuldt tilgængelige for alle.
"På trods af de resterende forhindringer, vores demonstration af, at en billig enhed pålideligt kan læse sekvenserne af naturligt forekommende DNA og kan fortolke DNA-segmenter så længe som 4, 500 nukleotider i længden repræsenterer et stort fremskridt inden for nanopore DNA-sekventering, " sagde Gundlach.
Sidste artikelDiamantplader skaber nanostrukturer gennem tryk, ikke kemi
Næste artikelSe væsker i nanoskala strømme
 Varme artikler
Varme artikler
-
 Vibrationsenergi hemmeligheden bag selvdrevet elektronik(Phys.org)-Et multi-universitetsteam af ingeniører har udviklet, hvad der kunne være en lovende løsning til opladning af smartphone-batterier på farten-uden behov for en elektrisk ledning. Indbygget
Vibrationsenergi hemmeligheden bag selvdrevet elektronik(Phys.org)-Et multi-universitetsteam af ingeniører har udviklet, hvad der kunne være en lovende løsning til opladning af smartphone-batterier på farten-uden behov for en elektrisk ledning. Indbygget -
 Ultraviolet til at producere ultratynd i ny finansieringForskere ved University of Liverpool undersøger brugen af ultraviolet lys til at skabe tynde film, der kan bruges på solpaneler, og at holde mad og medicinske instrumenter sterile. Finansiering fra
Ultraviolet til at producere ultratynd i ny finansieringForskere ved University of Liverpool undersøger brugen af ultraviolet lys til at skabe tynde film, der kan bruges på solpaneler, og at holde mad og medicinske instrumenter sterile. Finansiering fra -
 Ionisk termisk op-diffusion booster energihøst(a) Skematisk illustration af ionkoncentrationsprofilerne med asymmetriske transmembrane temperaturforskelle. (b) Elektrisk energi under forskellige temperaturforskelle. (c) Ved små transmembrane konc
Ionisk termisk op-diffusion booster energihøst(a) Skematisk illustration af ionkoncentrationsprofilerne med asymmetriske transmembrane temperaturforskelle. (b) Elektrisk energi under forskellige temperaturforskelle. (c) Ved små transmembrane konc -
 Forskere bruger lipid-nanopartikler til præcist at målrette genredigering til leverenForskere udviklede nanopartikler, der bærer en genredigeringspakke specifikt til leveren hos mus, med succes at reducere niveauet af LDL dårligt kolesterol i blodet. Sammen med tidligere undersøgelser
Forskere bruger lipid-nanopartikler til præcist at målrette genredigering til leverenForskere udviklede nanopartikler, der bærer en genredigeringspakke specifikt til leveren hos mus, med succes at reducere niveauet af LDL dårligt kolesterol i blodet. Sammen med tidligere undersøgelser
- En ud af tre floder på den iberiske halvø er påvirket af tilsaltning
- Sådan konverteres højde til tommer på et diagram
- Blå skraldespande flyder over med Amazon- og Walmart -kasser. Men var faktisk ved at genbruge mindr…
- Lokkeeffekten:hvordan du bliver påvirket til at vælge uden rigtig at vide det
- Identifikation af designermedicin taget af overdosispatienter
- Vi introducerer mesh, et hukommelsesbesparende plug-in, der kan øge telefonens og computerens ydeev…


