Løsning af labyrinter med enkelt-molekyle DNA-navigatorer
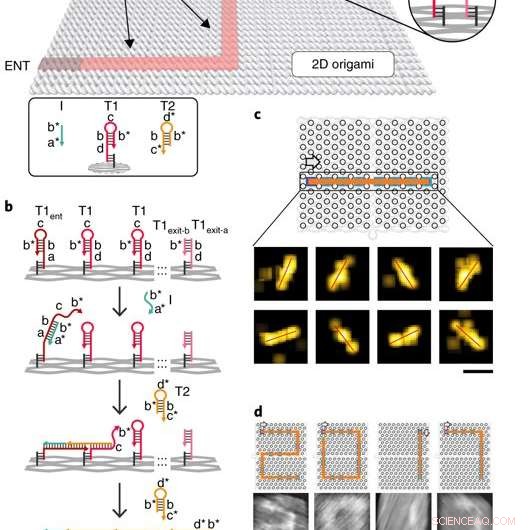
Implementering af enkelt-molekyle DNA-navigator. a) Skematisk illustration af PSEC-systemet. En acyklisk forbundet graf (dvs. et træ) er konstrueret på et rektangulært DNA-origami-substrat på 100 x 70 nm^2. T1 og T2 er to typer brændstoffer, der driver PSEC på træet. Initiator I blev brugt til at udløse initieringen af PSEC fra indgangspunktet ENT. b) PSEC-systemets udbredelsesmekanisme. Initiator I genkender og åbner T1ent-hårnålen ved vertex ENT. Den åbnede hårnål fanger derefter og åbner en T2 hårnål fra omgivelserne for at starte kaskaden. c) En lige linje brolagt af PSEC og visualiseret med DNA-PAINT. d) Fire brolagte cifre af "2017" afbildet ved hjælp af atomkraftmikroskopi (AFM). T1-hårnålene ved hjørnepunkterne blev modificeret for at forhindre uønskede spænd. Pilene angiver udbredelsesretningen. Kredit: Naturmaterialer , doi:10.1038/s41563-018-0205-3.
Feltet for intelligent nanorobotik er baseret på det store løfte om molekylære enheder med informationsbehandlingskapacitet. I en ny undersøgelse, der understøtter tendensen med DNA-baserede informationsbærere, forskere har konstrueret et DNA-navigatorsystem, der kan udføre enkelt-molekyle, parallel, dybde-første søgeoperationer på en todimensionel origami-platform.
Pathfinding-operationer med DNA-navigatorer gør brug af en lokaliseret strengudvekslingskaskadeproces, der initieres på et unikt triggersted på origami-platformen. Automatisk progression langs stier aktiveres af DNA-hårnåle, der indeholder en universel gennemløbssekvens. Af design, hver enkelt-molekyle navigator kan selvstændigt udforske enhver af de mulige stier gennem et 10-vertex rodfæstet træ konstrueret i undersøgelsen. Labyrinterne svarede til et træ med en indgang ved roden og en udgang gennem et af bladene. Undersøgelsen udført af Jie Chao og kolleger resulterede i at udforske alle stier, som DNA-navigatorerne tog for at udtrække en specifik løsningssti, der forbandt et givent par start- og slutspidser i labyrinten. Som resultat, opløsningsvejen blev lagt tydeligt på origami-platformen og illustreret ved hjælp af enkelt-molekyle billeddannelse. Fremgangsmåden er nu offentliggjort i Naturmaterialer , detaljering af realiseringen af molekylære materialer med indlejrede biomolekylære beregningsfunktioner til at fungere på niveau med enkeltmolekylet med potentiale til at konstruere intelligente nanorobotter til fremtidige applikationer inden for industri og medicin.
Sofistikerede molekylære værktøjer blev tidligere brugt til at skabe molekylære maskiner, der omdanner kemikalier, fotonisk eller elektrisk energi til roterende eller lineære bevægelser på nanoskala. For eksempel, Brownsk bevægelse på nanoskalaen kan kontrolleres omdannes til rettede bevægelser i DNA-baserede nanomaskiner ved hjælp af DNA-hybridiseringsreaktioner. Sådanne DNA-baserede maskiner fungerer autonomt ved at følge et indlejret 'molekylært program', der er foruddesignet som en kaskadereaktion, der manuelt udløses via en ekstern stimulus for hvert trin af operationen.
Fokus på feltet er gradvist skiftet til at aktualisere DNA-baserede logiske kredsløb ved hjælp af aptamerer og DNAzymer til at designe molekylære logiske porte. For eksempel, i 2006, Stojanovic og hans kolleger integrerede mere end 100 DNA-logiske porte for at udvikle en automatisering kaldet MAYA-II for at spille et spil Tic-Tac-Toe. Tidligere undersøgelser viste et enzymfrit computersystem baseret på hybridiseringskædereaktioner (HCR) for at skabe logiske porte og logiske kredsløb for mere robust og effektiv ydeevne end de originale systemer.
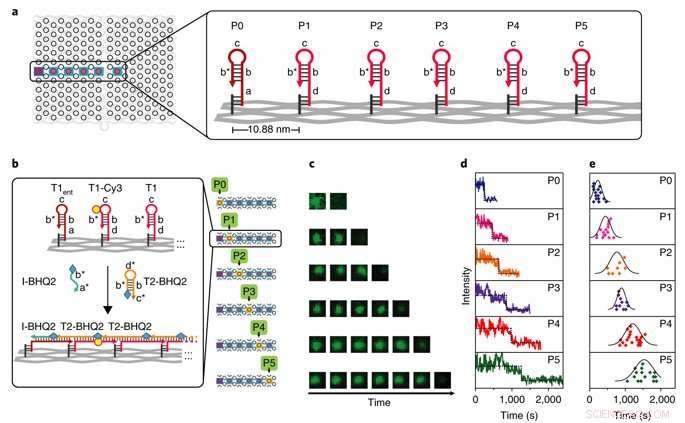
Enkeltmolekyle karakterisering af PSEC kinetik. a) En lige linje i midten af DNA-origamien blev brugt som testleje. Toppunkt P0 er udgangspunktet og toppunkter P1-P5 er mellempunkter. b) Detaljer om designet af seks parallelle test til måling af kinetikken med tidsopløst TIRF. T2 mærket med BHQ2 blev brugt til at slukke T1 mærket med Cy3, samle en kinetisk profil ved hvert trin. Det illustrerede eksempel viser slukningsmekanismen. c) Eksempel på TIRF-billeder viser, at fluorescensen ændrer sig med tiden over de seks parallelle tests fra P0 til P5. d) Typiske enkeltmolekyle fluorescensspor, der anvendes til at overvåge quenching-hændelser, der forekommer ved toppunkterne P0 til P5. e) Spredningsplot, der viser statistisk analyse af længdefordelingen pr. vej. Kredit: Naturmaterialer , doi:10.1038/s41563-018-0205-3.
I denne undersøgelse af Chao et al. det samme grundlæggende princip i HCR-reaktionsskemaet blev brugt i en anden beregningsmæssig sammenhæng til at udvikle et enkelt-molekyle DNA-navigatorsystem. Platformen udforskede alle mulige stier gennem en trægraf designet på en origami-struktur som en simpelt forbundet labyrint uden cykliske stier. Sådanne DNA-origamistrukturer er informationsbærende nanostrukturer af natur med veldefineret nanoskala geometri. Labyrinten kunne udforskes ved hjælp af proksimal strengudvekslingskaskade (PSEC) baseret på hybridiseringskædereaktioner. Forskerne demonstrerede, at et system med et stort antal enkeltmolekylære DNA-navigatorer i fællesskab kunne udføre parallel dybde-først-søgning (PDFS) på træet for effektivt at udføre labyrintløsning inden for 2-D origami. I første omgang gennemførte forskerne undersøgelser for at teste PSEC-designet.
Det proksimale strengudvekslingskaskadesystem (PSEC) (arbejdsprincippet for DNA-navigatoren) blev faciliteret på et rektangulært origami-substrat lavet af tre komponenter, som omfattede den fysiske implementering af en trægraf, fulde strenge og en initiatorstreng. Ledige områder uden hæfteforlængelser svarede til vægge i labyrinten, forhindrer udbredelse af strengudvekslingskaskaden. The entrance and exit were defined and denoted as ENT and EXIT respectively. In the second component, two types of DNA hairpins, T1 and T2, were used as fuels to drive the PSEC on the tree graph. The two hairpins coexisted metastably in solution to hybridize and fuel the PSEC process with free energy .
Af design, information only propagated through the network in the presence of an initiator (Initiator I). Upon addition of initiator I, PSEC was conducted and observed using atomic force microscopy (AFM). To visualize an established formation, the scientists enabled DNA-navigator-based formation of the number 2017 as a proof-of-principle. Another technique known as DNA-PAINT was employed as a single-molecule, super-resolution imaging technique to reveal molecular features at the nanoscale to further substantiate the PSEC-based path paving process. The on-origami PSEC was highly specific, without intra- or inter-origami crosstalk.
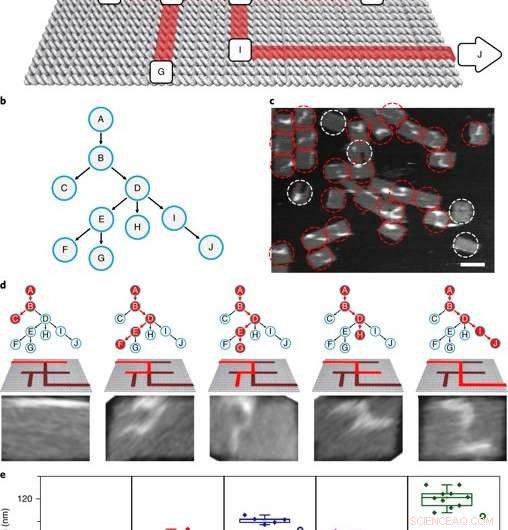
The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Only P ABDIJ was the correct solution to the maze. e) Scatter plots showing the statistical analysis of the length distribution for each path. Kredit: Naturmaterialer , doi:10.1038/s41563-018-0205-3.
Kinetics of the process were investigated at the single-molecule level in the study using time-resolved total internal reflection fluorescence microscopy (TIRF) in a setup with a prescribed starting point (P0) and five intermediate steps (P1-P5). Fluorescence in the setup was quenched using fluorescence resonance energy transfer (FRET), and the cascade was observed in real time by recording fluorescence signals continuously. The average speed of propagation was recorded to be 2.46 nm per minute, propagation across the straight line (54.4 nm) took approximately 22 minutes.
The scientists then constructed the main model maze with 10 vertices that included an entrance vertex A and an exit vertex J, three junctions (B, D, E) an intermediate vertex (I) and four dead ends (C, F, G, H). Each path of the maze that was equivalent to a tree with 10 vertices was investigated using DNA investigators starting at root A. The PSEC reactions produced a mixture of various paths on the maze, confirmed with AFM. Each individual PSEC could progress on one of the five possible paths. Statistical analysis of the length distribution showed that the measured paths coincided well with the values predicted.
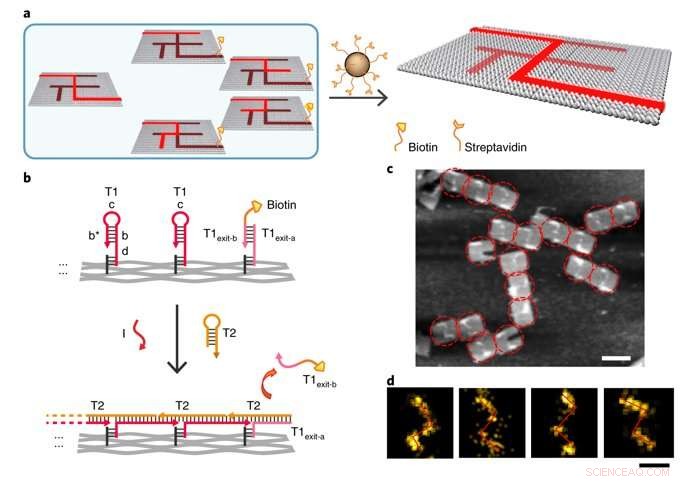
Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Kredit: Naturmaterialer , doi:10.1038/s41563-018-0205-3.
To prevent the navigators from propagating through a wrong path with dead ends, the scientists designed a streptavidin-biotin tag-based method to selectively eliminate inaccurate path navigation. Only the correct path (P ABDIJ ) was followed in the maze therefore. The computational context used in the study allowed exploration of paths through tree graphs defined on the origami. The autonomous path explored by the DNA navigators proceeded unidirectionally and irreversibly, turning at junctions and corners on the origami platform as they were designed to. The design enabled parallel depth first search (PDFS) allowing each DNA navigator to individually explore any one of the paths through the given graph at a defined speed, greater than that previously achieved.
The main advantage of the described biomolecular computer schemes in comparison to conventional electronic computing is they can be interfaced directly with biologically relevant processes. Som resultat, the scientists envision translational biomedical sensing and decision-making platforms with DNA origami and single-molecule diagnostics using decision trees. Future applications will also include simple sensors or those coupled to a molecular actuator to trigger downstream molecular cascades.
© 2018 Science X Network
 Varme artikler
Varme artikler
-
 Ingeniører udvikler nanoskum til bedre rustning, beskyttelseslag for bygningerFra venstre:Porøs silica, med en gennemsnitlig porestørrelse på nogle få mikron set ved 5-mikron, 20 mikron og 50 mikron skala. (Phys.org) — Ingeniører ved University of California, San Diego er v
Ingeniører udvikler nanoskum til bedre rustning, beskyttelseslag for bygningerFra venstre:Porøs silica, med en gennemsnitlig porestørrelse på nogle få mikron set ved 5-mikron, 20 mikron og 50 mikron skala. (Phys.org) — Ingeniører ved University of California, San Diego er v -
 Nanofotonik teknologi muliggør en ny slags optisk spektrometerDr. Junpeng Guo, UAHuntsville lektor i elektroteknik og optik, og ph.d.-studerende Haisheng Leong ser spektrene fra en ny fotonisk enhed i nanoskala kaldet et supernano-rist. Kredit:Aaron Sexton / UAH
Nanofotonik teknologi muliggør en ny slags optisk spektrometerDr. Junpeng Guo, UAHuntsville lektor i elektroteknik og optik, og ph.d.-studerende Haisheng Leong ser spektrene fra en ny fotonisk enhed i nanoskala kaldet et supernano-rist. Kredit:Aaron Sexton / UAH -
 Nanopartikler kan udnytte tumorsvagheder til selektivt at angribe kræftformerNanopartiklerne er belagt med et lag polymer, der maskerer den fra genkendelse og eliminering af immunsystemet, når den cirkulerer i kroppen. Når det når en tumor, det frigiver langsomt et lægemiddelp
Nanopartikler kan udnytte tumorsvagheder til selektivt at angribe kræftformerNanopartiklerne er belagt med et lag polymer, der maskerer den fra genkendelse og eliminering af immunsystemet, når den cirkulerer i kroppen. Når det når en tumor, det frigiver langsomt et lægemiddelp -
 Ny indsigt i grafen og organiske kompositter i elektronikSkematisk, der repræsenterer organiske molekyler knyttet til grafen, svagt interagerende med 2d-materialet gennem supramolekylære interaktioner. Kredit:© 2015 Vincenzo Palermo/CNR-ISOF Kemikere fr
Ny indsigt i grafen og organiske kompositter i elektronikSkematisk, der repræsenterer organiske molekyler knyttet til grafen, svagt interagerende med 2d-materialet gennem supramolekylære interaktioner. Kredit:© 2015 Vincenzo Palermo/CNR-ISOF Kemikere fr
- Videnskabelige undersøgelser af troede rester af to apostle
- Beregning af fordelene ved exascale og kvantecomputere
- Verden har aldrig set en kategori 6-orkan, men dagen kan komme
- Forskning nulstilling på elektronisk næse til overvågning af luftkvalitet, diagnosticering af syg…
- Sådan bestemmes lineære ligninger
- Oceanografer løser mysteriet om fytoplanktons overlevelse i det næringsfattige Stillehav


