Forskere kortlægger det menneskelige genom i 4-D, når det folder sig
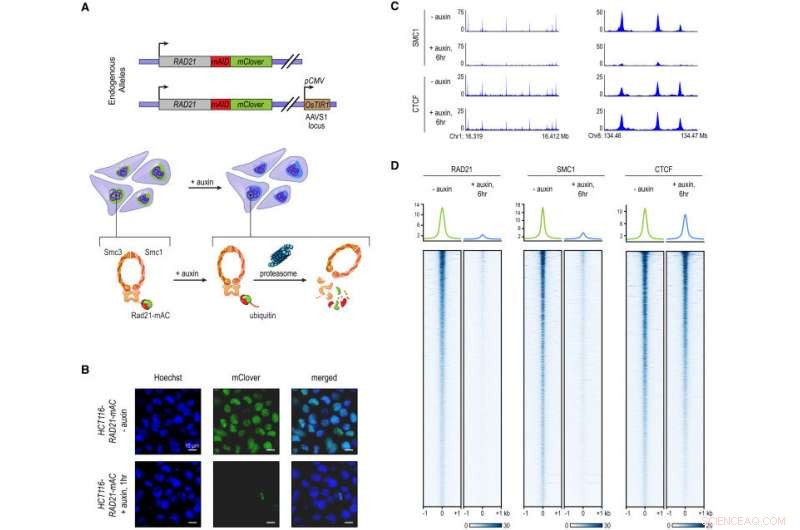
Mærkning af endogen RAD21 med en auxin-inducerbar Degron muliggør hurtig, Nær komplet tab af Cohesin. (A) I HCT-116-RAD21-mAC celler, begge RAD21-alleler er mærket med auxin-inducerbare degroner og en mClover-reporter, og OsTIR1 genet er integreret på AAVS1 locus. Auxinbehandling fører til proteasomal nedbrydning af RAD21. (B) Live cell imaging efter Hoechst 33342 farvning til mærkning af kerner. Nuklear mClover -fluorescens svarende til mærket RAD21 gik tabt efter 1 times auxinbehandling. (C) SMC1 og CTCF ChIP-seq signal med og uden auxinbehandling. (D) RAD21, SMC1, og CTCF ChIP-seq signal (venstre, midten, til højre) på tværs af alle toppe kaldte til hvert af proteinerne i ubehandlede RAD21-mAC-celler. Top:Gennemsnitlige berigelser for hvert protein. Efter nedbrydning af RAD21, cohesin -komplekset binder ikke længere til kromatin. CTCF -binding påvirkes ikke. Kredit: Celle . DOI:10.1016/j.cell.2017.09.026
Et multiinstitutionelt team, der spænder over Baylor College of Medicine, Rice University, Stanford University og Broad Institute of MIT og Harvard har skabt det første højopløselige 4-D-kort over genomfoldning, spore et helt menneskeligt genom, når det folder sig over tid. Rapporten, som kan føre til nye måder at forstå genetiske sygdomme på, vises på forsiden af Celle .
Oprettelse af forbindelser
I årtier, forskere har mistanke om, at når en menneskelig celle reagerer på en stimulus, DNA -elementer, der ligger langt fra hinanden i genomet, finder hurtigt hinanden, danner sløjfer langs kromosomet. Ved at omarrangere disse DNA -elementer i rummet, cellen er i stand til at ændre, hvilke gener der er aktive.
I 2014, det samme team af forskere viste, at det var muligt at kortlægge disse sløjfer. Men de første kort var statiske, uden evnen til at se sløjferne ændre sig. Det var uklart, om i det overfyldte rum i kernen, DNA -elementer kunne finde hinanden hurtigt nok til at kontrollere cellulære reaktioner.
"Før, vi kunne lave kort over, hvordan genomet foldede sig, når det var i en bestemt tilstand, men problemet med et statisk billede er, at hvis intet nogensinde ændrer sig, det er svært at finde ud af, hvordan tingene fungerer, "sagde Suhas Rao, første forfatter til den nye undersøgelse. "Vores nuværende tilgang er mere som at lave en film; vi kan se folder, når de forsvinder og dukker op igen."
Én ring for at styre dem alle
For at spore foldeprocessen over tid, forskergruppen begyndte med at forstyrre cohesin, et ringformet proteinkompleks, der var placeret ved grænserne for næsten alle kendte sløjfer. I 2015, teamet foreslog, at cohesin skaber DNA -sløjfer i cellekernen ved en ekstruderingsproces.
"Ekstrudering fungerer som justeringen af remlængden på en rygsæk, "forklarede Dr. Erez Lieberman Aiden, direktør for Center for Genome Architecture ved Baylor College of Medicine og seniorforfatter på det nye studie. "Når du fodrer remmen gennem hver side, det danner en sløjfe. DNA ser ud til at gøre det samme - bortset fra at kohesinringe ser ud til at spille justeringsrollen. "
Aiden sagde, at en afgørende forudsigelse af 2015 -modellen er, at alle sløjfer skal forsvinde i mangel af kohesin. I den nye forskning, Aiden, Rao og kolleger testede denne antagelse.
"Vi fandt ud af, at da vi forstyrrede samhørigheden, tusinder af sløjfer forsvandt, "sagde Rao, en lægestuderende ved Stanford University, Hertz Fellow og medlem af Aiden lab. "Derefter, når vi sætter cohesin tilbage, alle disse sløjfer kom tilbage - ofte i løbet af få minutter. Dette er præcis, hvad du ville forudsige fra ekstruderingsmodellen, og det tyder på, at den hastighed, hvormed cohesin bevæger sig langs DNA, er blandt de hurtigste for et kendt humant protein. "
Sløjfer kontra grupper
Men alt skete ikke, som forskerne forventede. I nogle tilfælde, sløjfer gjorde det stik modsatte af, hvad forskerne forventede.
"Da vi så tusindvis af sløjfer på tværs af genomet blive svagere, vi bemærkede et sjovt mønster, "sagde Aiden, også en McNair Scholar, Hertz Fellow og en seniorforsker ved Rice Universitys Center for Teoretisk Biologisk Fysik. "Der var et par ulige sløjfer, der faktisk blev stærkere. Derefter, da vi satte cohesin tilbage, de fleste sløjfer kom sig fuldstændigt - men disse ulige sløjfer gjorde igen det modsatte - de forsvandt! "
Ved at undersøge, hvordan kortene ændrede sig over tid, teamet indså, at ekstrudering ikke var den eneste mekanisme, der bringer DNA -elementer sammen. En anden mekanisme, kaldes opdeling, involverede ikke cohesin.
"Den anden mekanisme, vi observerede, er ganske anderledes end ekstrudering, "forklarede Rao." Ekstrudering har en tendens til at bringe DNA -elementer sammen to ad gangen, og kun hvis de ligger på det samme kromosom. Denne anden mekanisme kan forbinde store grupper af elementer med hinanden, selvom de ligger på forskellige kromosomer. Og det ser ud til at være lige så hurtigt som ekstrudering. "
Broad Institute Director Eric Lander, en medforfatter af et studie, sagde, "Vi begynder at forstå de regler, hvormed DNA -elementer samles i kernen. Nu hvor vi kan spore elementerne, når de bevæger sig over tid, de underliggende mekanismer begynder at blive tydeligere. "
Sidste artikelKulminearbejdere skifter til biavl
Næste artikelRådyr foretrækker indfødte planter, der efterlader varig skade på skovene
 Varme artikler
Varme artikler
-
 Forstå sproget i mobilkommunikationI flercellede organismer, celler skal adressere beskeder til specifikke andre celletyper. For at gøre det, celler bruger et sæt lignende, men ikke identisk, signalerende proteiner, eller ligander. Hve
Forstå sproget i mobilkommunikationI flercellede organismer, celler skal adressere beskeder til specifikke andre celletyper. For at gøre det, celler bruger et sæt lignende, men ikke identisk, signalerende proteiner, eller ligander. Hve -
 Uventet fund i cellens kraftværkMitokondrier. Kredit:Wikipedia commons Forskere ved Karolinska Institutet har opdaget, at proteinkomplekset RNase P i cellens mitokondrier opfører sig anderledes end hidtil antaget. Fundene, udgiv
Uventet fund i cellens kraftværkMitokondrier. Kredit:Wikipedia commons Forskere ved Karolinska Institutet har opdaget, at proteinkomplekset RNase P i cellens mitokondrier opfører sig anderledes end hidtil antaget. Fundene, udgiv -
 At spejde ørnene:Bevis på, at beskyttelse af reder hjælper reproduktionenSkaldede ørne har en tendens til at rede langs de mange søbredder i Voyageurs National Park, Minnesota. Kredit:Jennyffer Cruz Reproduktion blandt skaldede ørne i en fjerntliggende nationalpark i M
At spejde ørnene:Bevis på, at beskyttelse af reder hjælper reproduktionenSkaldede ørne har en tendens til at rede langs de mange søbredder i Voyageurs National Park, Minnesota. Kredit:Jennyffer Cruz Reproduktion blandt skaldede ørne i en fjerntliggende nationalpark i M -
 Eneboer eller ej? Forskere bruger Twitter til at løse edderkoppespørgsmålBrun eneboer (Loxosceles reclusa). Kredit:Matt Bertone Eneboer edderkopper, især den brune eneboer (Loxosceles reclusa), er en af verdens mest udskældte grupper af edderkopper. Alle tror, de k
Eneboer eller ej? Forskere bruger Twitter til at løse edderkoppespørgsmålBrun eneboer (Loxosceles reclusa). Kredit:Matt Bertone Eneboer edderkopper, især den brune eneboer (Loxosceles reclusa), er en af verdens mest udskældte grupper af edderkopper. Alle tror, de k


