Matematik gør det muligt for videnskabsmænd at forstå organisationen i en cellekerne
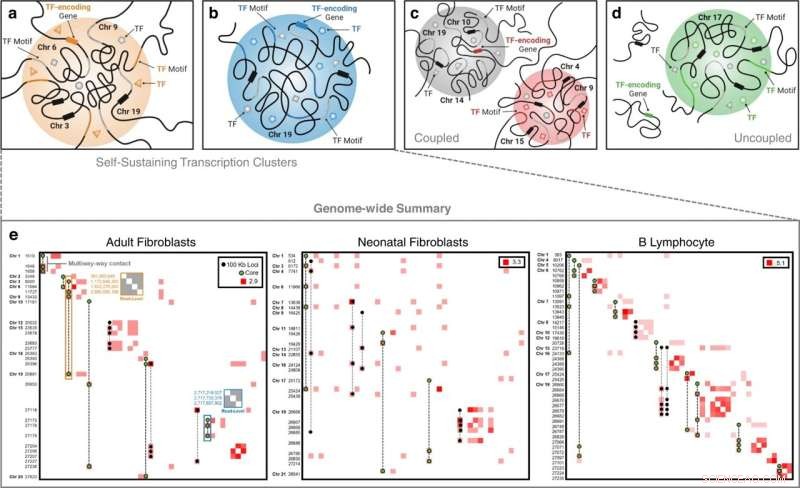
Klasser af transkriptionsklynger. I en selvbærende transkriptionsklynge er en TF og genet, der koder for den TF, begge til stede. De inter- og intrakromosomale eksempler i henholdsvis (a) og (b) illustrerer dette fænomen, hvor vi i a ser den TF af interesse (orange trekant) cirkulere ved klyngen, og dens bindingsmotiv er til stede på kromatinen (orange del). ), og dets tilsvarende gen udtrykt (orange rektangel på kromosom 6). De grå former repræsenterer yderligere TF'er med bindingsmotiver (grå del af kromatin) ved klyngen. Sorte rektangler på kromosom 3, 9 og 19 repræsenterer yderligere gener til stede i klyngen. c En analog-uafhængig klasse af transkriptionsklynger, hvor vi observerer en TF (rød firkant) binder til en transkriptionsklynge (rød klynge) og dets tilsvarende gen udtrykt i en separat transkriptionsklynge (grå klynge), men ikke i samme klynge. d En analog-uafhængig klasse af transkriptionsklynger, hvor vi observerer en TF (grøn cirkel) binder til en transkriptionsklynge (grøn klynge) og dets tilsvarende gen udtrykt, men ikke inden for en transkriptionsklynge. e Genomomfattende celletypespecifikke selvopretholdende transkriptionsklynger ekstraheret fra multi-vejs kontaktdata og dekomponeret i Hi-C kontaktmatricer ved 100 kb opløsning. Kontaktfrekvenser log-transformeres for bedre visualisering. Frekvenser langs diagonalen indikerer interaktion mellem to eller flere unikke multi-vejs loci, der falder inden for den samme 100 kb bin. Akselabels er ikke-sammenhængende 100 kb bin-koordinater i kromosomal rækkefølge. Flervejskontakter, der udgør de selvbærende transkriptionsklynger, er overlejret. Flervejskontakter med grønfarvede loci repræsenterer 'kerne' transkriptionsklynger - transkriptionsklynger, der indeholder en masterregulator og dens genanalog. Et eksempel på læseniveau-kontaktkort for den inter-kromosomale FOXO3-selvbærende transkriptionsklynge er angivet med den orange fremhævede boks i den voksne fibroblastkontaktmatrix og et læseniveau-kontaktkort for den intrakromosomale ZNF320 selvbærende transkriptionsklynge er angivet med den blå fremhævede boks. Værdier langs venstre akse af disse læseniveau-kontaktmatricer er basepar-positioner af de kontaktende loci i genomet. Kredit:Nature Communications (2022). DOI:10.1038/s41467-022-32980-z
Science fiction-forfatteren Arthur C. Clarkes tredje lov siger, at "enhver tilstrækkeligt avanceret teknologi kan ikke skelnes fra magi."
Indika Rajapakse, Ph.D., er troende. Ingeniøren og matematikeren er nu biolog. Og han mener, at skønheden ved at blande disse tre discipliner er afgørende for at optrevle, hvordan celler fungerer.
Hans seneste udvikling er en ny matematisk teknik til at begynde at forstå, hvordan en celles kerne er organiseret. Teknikken, som Rajapakse og samarbejdspartnere testede på flere typer celler, afslørede, hvad forskerne kaldte selvbærende transkriptionsklynger, en undergruppe af proteiner, der spiller en nøglerolle i at opretholde celleidentitet.
De håber, at denne forståelse vil afsløre sårbarheder, der kan målrettes for at omprogrammere en celle til at stoppe kræft eller andre sygdomme.
"Flere og flere kræftbiologer mener, at genomorganisering spiller en enorm rolle i forståelsen af ukontrollerbar celledeling, og om vi kan omprogrammere en kræftcelle. Det betyder, at vi er nødt til at forstå flere detaljer om, hvad der sker i kernen," siger Rajapakse, lektor i computational. medicin og bioinformatik, matematik og biomedicinsk teknik ved University of Michigan. Han er også medlem af U-M Rogel Cancer Center.
Rajapakse er seniorforfatter på papiret, udgivet i Nature Communications . Projektet blev ledet af en trio af kandidatstuderende med et tværfagligt team af forskere.
Holdet forbedrede en ældre teknologi til at undersøge kromatin, kaldet Hi-C, der kortlægger hvilke dele af genomet, der er tæt på hinanden. Det kan identificere kromosomtranslokationer, som dem der forekommer i nogle kræftformer. Dens begrænsning er imidlertid, at den kun ser disse tilstødende genomiske regioner.
Den nye teknologi, kaldet Pore-C, bruger meget flere data til at visualisere, hvordan alle dele i en celles kerne interagerer. Forskerne brugte en matematisk teknik kaldet hypergrafer. Tænk:tredimensionelt Venn-diagram. Det giver forskerne mulighed for at se ikke kun par af genomiske regioner, der interagerer, men helheden af de komplekse og overlappende genom-dækkende relationer i cellerne.
"Dette multi-dimensionelle forhold kan vi forstå utvetydigt. Det giver os en mere detaljeret måde at forstå organisatoriske principper inde i kernen. Hvis du forstår det, kan du også forstå, hvor disse organisatoriske principper afviger, som i kræft," sagde Rajapakse. "Dette er som at sætte tre verdener sammen - teknologi, matematik og biologi - for at studere flere detaljer inde i kernen."
Forskerne testede deres tilgang på neonatale fibroblaster, biopsierede voksne fibroblaster og B-lymfocytter. De identificerede organisationer af transkriptionsklynger, der er specifikke for hver celletype. De fandt også, hvad de kaldte selvbærende transkriptionsklynger, som tjener som nøgletransskriptionssignaturer for en celletype.
Rajapakse beskriver dette som det første skridt i et større billede.
"Mit mål er at konstruere denne slags billede over cellecyklussen for at forstå, hvordan en celle går gennem forskellige stadier. Kræft er ukontrollerbar celledeling," sagde Rajapakse. "Hvis vi forstår, hvordan en normal celle ændrer sig over tid, kan vi begynde at undersøge kontrollerede og ukontrollerede systemer og finde måder at omprogrammere det system på." + Udforsk yderligere
Afsløring af mysterierne bag genomstrukturen i den menneskelige cellekerne ved hjælp af en 3D-beregningssimulering
 Varme artikler
Varme artikler
-
 Videnskabsprojekt om hvorfor knogler får gummi i eddikeGummi knogleeksperimentet er en klassisk videnskabelig undersøgelse, der lærer hvor vigtigt calcium er for at have et stærkt og sundt skeletsystem samt de sure egenskaber ved eddike. Du kan udføre det
Videnskabsprojekt om hvorfor knogler får gummi i eddikeGummi knogleeksperimentet er en klassisk videnskabelig undersøgelse, der lærer hvor vigtigt calcium er for at have et stærkt og sundt skeletsystem samt de sure egenskaber ved eddike. Du kan udføre det -
 Tigre klynger sig til overlevelse i Sumatras stadig mere fragmenterede skoveEn anden tiger poserer for en kamerafælde på Sumatra. Kredit:Matthew Luskin En forskningsekspedition sporede truede tigre gennem Sumatran-junglen i et år og fandt, at tigre klynger sig til overlev
Tigre klynger sig til overlevelse i Sumatras stadig mere fragmenterede skoveEn anden tiger poserer for en kamerafælde på Sumatra. Kredit:Matthew Luskin En forskningsekspedition sporede truede tigre gennem Sumatran-junglen i et år og fandt, at tigre klynger sig til overlev -
 Forholdet mellem myosit i eukaryotiske celler og binær fission i prokaryoterCellerne i alle levende organismer har karakteristika i almindelighed såvel som mange forskelle. En fællesitet er evnen til at vokse og reproducere gennem produktion af nye celler; celledeling. Binær
Forholdet mellem myosit i eukaryotiske celler og binær fission i prokaryoterCellerne i alle levende organismer har karakteristika i almindelighed såvel som mange forskelle. En fællesitet er evnen til at vokse og reproducere gennem produktion af nye celler; celledeling. Binær -
 Fordelene ved at bruge klæbrige enzymerMolekylær kloning er en almindelig bioteknologisk metode, som enhver studerende og forsker skal være bekendt med. Molekylær kloning ved anvendelse af en type enzym kaldet et restriktionsenzym til a
Fordelene ved at bruge klæbrige enzymerMolekylær kloning er en almindelig bioteknologisk metode, som enhver studerende og forsker skal være bekendt med. Molekylær kloning ved anvendelse af en type enzym kaldet et restriktionsenzym til a
- Ultrahøj termisk isolering på tværs af heterogent lagdelte todimensionelle materialer
- Kvanteprikker sporer, hvem der kommer ind i cellekernen
- Chancerne for livet udvides, når forbipasserende stjerner skubber binære filer sammen
- National undersøgelse viser, at rekreation mellem hav og kyst er en stor forretning
- Følelse af nanoskalaen med synligt lys, og det grundlæggende ved uordnede bølger
- Ny optisk metode baner vej for vejrtrækningstest for kræftbiomarkør


