Beyond AlphaFold:AI udmærker sig ved at skabe nye proteiner

Proteiner designet med et ultrahurtigt softwareværktøj kaldet ProteinMPNN var meget mere tilbøjelige til at folde op efter hensigten. Kredit:Ian Haydon, UW Medicine Institute for Protein Design
I løbet af de sidste to år har maskinlæring revolutioneret forudsigelse af proteinstruktur. Nu, tre artikler i Science beskrive en lignende revolution inden for proteindesign.
I de nye artikler viser biologer ved University of Washington School of Medicine, at maskinlæring kan bruges til at skabe proteinmolekyler meget mere præcist og hurtigere end tidligere muligt. Forskerne håber, at dette fremskridt vil føre til mange nye vacciner, behandlinger, værktøjer til kulstoffangst og bæredygtige biomaterialer.
"Proteiner er fundamentale på tværs af biologien, men vi ved, at alle de proteiner, der findes i hver plante, dyr og mikrobe, udgør langt mindre end én procent af, hvad der er muligt. Med disse nye softwareværktøjer burde forskere være i stand til at finde løsninger på lang -stående udfordringer inden for medicin, energi og teknologi," sagde seniorforfatter David Baker, professor i biokemi ved University of Washington School of Medicine og modtager af en 2021 Breakthrough Prize in Life Sciences.
Proteiner omtales ofte som "livets byggesten", fordi de er essentielle for alle levende tings struktur og funktion. De er involveret i stort set alle processer, der finder sted inde i celler, inklusive vækst, deling og reparation. Proteiner er opbygget af lange kæder af kemikalier kaldet aminosyrer. Rækkefølgen af aminosyrer i et protein bestemmer dets tredimensionelle form. Denne indviklede form er afgørende for, at proteinet kan fungere.
For nylig er kraftfulde maskinlæringsalgoritmer inklusive AlphaFold og RoseTTAFold blevet trænet til at forudsige de detaljerede former for naturlige proteiner udelukkende baseret på deres aminosyresekvenser. Machine learning er en form for kunstig intelligens, der gør det muligt for computere at lære af data uden at være eksplicit programmeret. Maskinlæring kan bruges til at modellere komplekse videnskabelige problemer, som er for svære for mennesker at forstå.
For at gå ud over de proteiner, der findes i naturen, opdelte Bakers teammedlemmer udfordringen med proteindesign i tre dele og brugte nye softwareløsninger til hver.
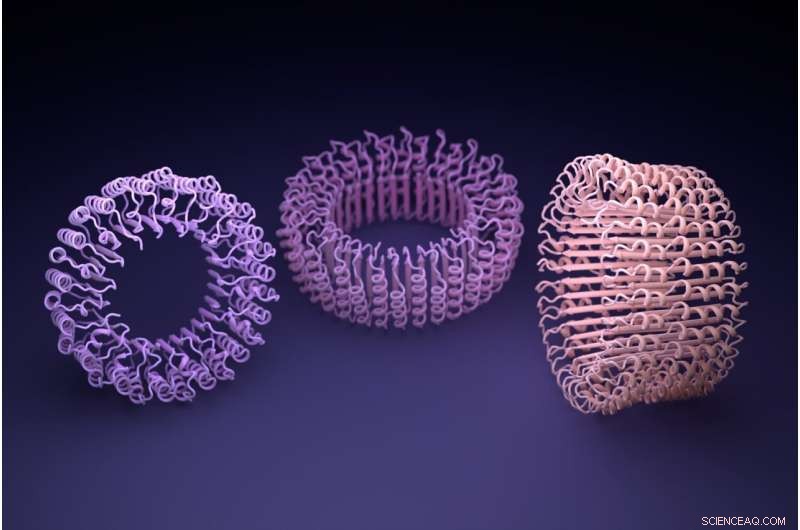
Kunstig intelligens hallucinerede disse symmetriske proteinsamlinger på en måde, der ligner andre A.!. generative værktøjer, der producerer output baseret på simple prompter. Kredit:Ian Haydon, UW Medicine Institute for Protein Design
Først skal der genereres en ny proteinform. I et papir offentliggjort 21. juli i tidsskriftet Science , viste holdet, at kunstig intelligens kan generere nye proteinformer på to måder. Den første, kaldet "hallucination", er beslægtet med DALL-E eller anden generativ A.I. værktøjer, der producerer output baseret på simple prompter. Den anden, kaldet "inpainting", er analog med autofuldførelsesfunktionen, der findes i moderne søgebjælker.
For det andet, for at fremskynde processen, udtænkte holdet en ny algoritme til generering af aminosyresekvenser. Beskrevet i udgaven af 15. september af Science , kører dette softwareværktøj, kaldet ProteinMPNN, på cirka et sekund. Det er mere end 200 gange hurtigere end den tidligere bedste software. Dens resultater er bedre end tidligere værktøjer, og softwaren kræver ingen eksperttilpasning for at køre.
"Neurale netværk er nemme at træne, hvis man har et væld af data, men med proteiner har vi ikke så mange eksempler, som vi gerne ville. Vi skulle ind og identificere, hvilke egenskaber i disse molekyler der er de vigtigste. Det var lidt af forsøg og fejl," sagde projektforsker Justas Dauparas, en postdoktor ved Institute for Protein Design
For det tredje brugte holdet AlphaFold, et værktøj udviklet af Alphabet's DeepMind, til uafhængigt at vurdere, om de aminosyresekvenser, de fandt på, sandsynligvis ville foldes til de tilsigtede former.
"Software til at forudsige proteinstrukturer er en del af løsningen, men det kan ikke komme med noget nyt alene," forklarede Dauparas.
"ProteinMPNN er at proteindesigne, hvad AlphaFold var for forudsigelse af proteinstruktur," tilføjede Baker.

Detalje af et protein designet ved hjælp af et hurtigt værktøj kaldet ProteinMPNN, endnu et fremskridt i brugen af kunstig intelligens og maskinlæring i proteindesign. Kredit:Ian Haydon, UW Medicine Institute for Protein Design
I et andet papir, der vises i Science 15. september bekræftede et team fra Baker-laboratoriet, at kombinationen af nye maskinlæringsværktøjer pålideligt kunne generere nye proteiner, der fungerede i laboratoriet.
"Vi fandt ud af, at proteiner fremstillet ved hjælp af ProteinMPNN var meget mere tilbøjelige til at folde op efter hensigten, og vi kunne skabe meget komplekse proteinsamlinger ved hjælp af disse metoder," sagde projektforsker Basile Wicky, en postdoc-stipendiat ved Institute for Protein Design.
Blandt de nye proteiner, der blev lavet, var ringe i nanoskala, som forskerne mener kunne blive dele til specialfremstillede nanomaskiner. Elektronmikroskoper blev brugt til at observere ringene, som har diametre, der er omkring en milliard gange mindre end et valmuefrø.
"Dette er begyndelsen på maskinlæring i proteindesign. I de kommende måneder vil vi arbejde på at forbedre disse værktøjer for at skabe endnu mere dynamiske og funktionelle proteiner," sagde Baker.
Computerressourcer til dette arbejde blev doneret af Microsoft og Amazon Web Services. + Udforsk yderligere
Biologer træner kunstig intelligens til at generere medicin og vacciner
 Varme artikler
Varme artikler
-
 Generelle egenskaber ved ProtistaProtister er en unik gruppe af organismer, der omfatter et rige i klassificeringen af levende ting. De andre kongeriger er Eubacteria, Archaebacteria, Svampe, Planter og Dyr. Kongeriget Protista ind
Generelle egenskaber ved ProtistaProtister er en unik gruppe af organismer, der omfatter et rige i klassificeringen af levende ting. De andre kongeriger er Eubacteria, Archaebacteria, Svampe, Planter og Dyr. Kongeriget Protista ind -
 Klimapåvirkede ændringer i blomstring, frugt påvirker også fuglens overflod, aktiviteterLivscyklus ændres, såsom avl og smeltning, sammen med den overordnede artmængde viste sig at skifte i indfødte hawaiianske fugle, såsom iiwi, at falde sammen med klimarelaterede ændringer i den indfød
Klimapåvirkede ændringer i blomstring, frugt påvirker også fuglens overflod, aktiviteterLivscyklus ændres, såsom avl og smeltning, sammen med den overordnede artmængde viste sig at skifte i indfødte hawaiianske fugle, såsom iiwi, at falde sammen med klimarelaterede ændringer i den indfød -
 Dugongs vil sandsynligvis være funktionelt uddøde i KinaDugong i Ritchies-øgruppen, Neil Island, Andaman. Kredit:Vardhanjp/Wikimedia Commons, CC BY-SA 4.0 Et lille team af forskere fra Kina, Storbritannien og Grækenland rapporterer, at dugonger sandsynl
Dugongs vil sandsynligvis være funktionelt uddøde i KinaDugong i Ritchies-øgruppen, Neil Island, Andaman. Kredit:Vardhanjp/Wikimedia Commons, CC BY-SA 4.0 Et lille team af forskere fra Kina, Storbritannien og Grækenland rapporterer, at dugonger sandsynl -
 Hvis en art ikke har en stor nok genpulje,kan det stadig udvikle sig? Den skæggribbe står over for farerne ved en begrænset genpulje efter at være blevet jaget næsten til udryddelse. Se flere billeder af fugle. Franz Aberham/Workbook
Hvis en art ikke har en stor nok genpulje,kan det stadig udvikle sig? Den skæggribbe står over for farerne ved en begrænset genpulje efter at være blevet jaget næsten til udryddelse. Se flere billeder af fugle. Franz Aberham/Workbook
- Vejr og klima bruges i flæng. Det burde de ikke være
- Undersøgelse bidrager til produktion af fleksible elektroniske enheder
- Hvad hvis en hovedvandforsyning var inficeret med en eller anden form for bacte
- Grafen:Forskere finder ud af, hvordan man knuser verdens stærkeste materiale
- Nanoworld sneslyngemaskiner skærer lige kanaler i halvlederoverflader
- Ændrede en useriøs stjerne sammensætningen af vores solsystem?


