Kan proteiner huske det?
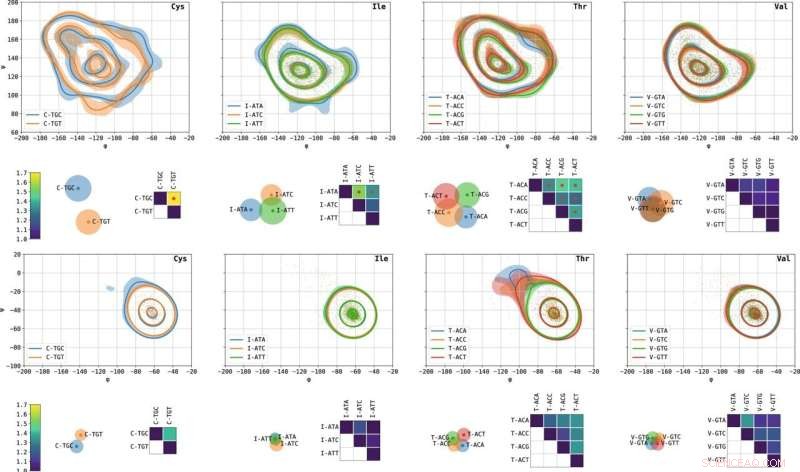
Kodonspecifikke Ramachandran-plot af udvalgte aminosyrer og afstande mellem dem. Vist fra venstre mod højre er cystein, isoleucin, threonin og valin. Konturplot viser niveaulinjerne, der indeholder 10, 50 og 90 % af sandsynlighedsmassen. Skraverede områder repræsenterer 10 %-90 % konfidensintervaller beregnet på 1000 tilfældige bootstraps. β- (øverst) og α- (nederst) tilstande er afbildet. Matricerne viser L1 afstande mellem par af kodonspecifikke Ramachandran-plot, normaliseret, så selvafstanden er 1. Røde prikker angiver par med signifikant forskellige dihedrale vinkelfordelinger baseret på deres p-værdi. Spredningsplottene, der visualiserer afstandsmatricerne, blev opnået ved en variant af multidimensionel skalering (MDS). Hvert punkt repræsenterer et kodon; parvise euklidiske afstande mellem punkterne tilnærmer L1 afstand mellem de tilsvarende kodoner. Cirkler tilnærmer usikkerhedsradierne. Jo mere to cirkler overlapper, jo mindre skelnelige er de tilsvarende kodonspecifikke Ramachandran-plot. Kredit:Nature Communications (2022). DOI:10.1038/s41467-022-30390-9
En undersøgelse, der integrerer biologiske ideer og nye computervidenskabelige værktøjer, har afsløret nye sammenhænge mellem genetisk kodning og proteinstruktur, som potentielt kan ændre den måde, vi tænker på proteinproduktion i ribosomet - cellens "protein samlebånd." Forskningen ledes af professor Alex Bronstein, Dr. Ailie Marx og Ph.D. studerende Aviv Rosenberg, blev udgivet i Nature Communications .
Proteiner, de komplekse molekyler, der spiller kritiske roller i stort set alle biologiske mekanismer, produceres af ribosomer i en proces kaldet translation. Ribosomet afkoder indkommende "genetiske instruktioner" for at syntetisere kæder af aminosyrer - proteinernes byggesten. Når aminosyrer er sekventielt bundet sammen til en lang kæde, foldes de til en unik tredimensionel struktur, der giver proteinet dets biologiske egenskaber og funktionalitet. Fejl i oversættelsen kan føre til fejlfoldning og efterfølgende fysiologiske lidelser, både milde og alvorlige.
Proteinproduktionsinstruktioner leveres til ribosomet som kodoner, sekvenser af tre "bogstaver" fra den genetiske nukleotidkode, som specificerer identiteten og rækkefølgen af aminosyrer, der skal tilføjes af ribosomet til proteinkæden. For eksempel signalerer codonet UUU til tilsætning af aminosyren phenylalanin, hvorimod codon UAC instruerer om tilsætning af tyrosin. På denne måde koder kodonsekvensen for den unikke sekvens af aminosyrer, der er karakteristiske for hvert protein. Denne kortlægning af genetiske kodoner til aminosyrer, der bruges til oversættelse, er fælles for alle levende væsner på planeten og betragtes som en urmekanisme.
Som om alt dette ikke var kompliceret nok, er det vigtigt at påpege, at der er 61 kodoner, der er afkodet til kun 20 aminosyrer. Med andre ord er alle undtagen to aminosyrer kodet af flere kodoner.
Det er her den nuværende forskning kommer ind i billedet. Baseret på eksperimenter udført i 1960'erne og 1970'erne, siger det accepterede dogme, at proteiner ikke bærer nogen "hukommelse" af det specifikke kodon, hvorfra hver aminosyre blev oversat, så længe aminosyreidentiteten forbliver uændret. Disse tidlige eksperimenter med proteinfoldning brugte kemiske denatureringsmidler til at udfolde fuldt dannede proteiner og viste derefter, at efter fjernelse af disse kemikalier kunne proteinkæden spontant foldes igen for at genvinde sin oprindelige struktur og funktion. Disse eksperimenter antydede, at kun aminosyresekvensen, og ikke den specifikke kodonsekvens, bestemmer et proteins struktur. I lyset af dette dogme bliver mutationer, der ændrer den genetiske kodning uden at ændre aminosyren, almindeligt betegnet som "tavse" og betragtes som uden betydning for proteinstruktur og funktion.
Forskerholdet fra Technion har afsløret en sammenhæng mellem identiteten af kodonet og den lokale struktur af det oversatte protein, hvilket tyder på, at dette muligvis ikke er det generelle tilfælde, og at proteiner faktisk kan "huske" de specifikke instruktioner, hvorfra de blev syntetiseret. Forskerholdet analyserede tusindvis af tredimensionelle proteinstrukturer ved hjælp af dedikerede værktøjer, de udviklede, som integrerer avancerede computervidenskabelige metoder, maskinlæring og statistik. På denne måde sammenlignede de nøjagtigt fordelingen af vinkler dannet i disse strukturer under forskellige synonyme genetiske koder. Deres resultater viser, at der for visse kodoner er en signifikant statistisk afhængighed mellem identiteten af kodonet og den lokale struktur af proteinet ved positionen af aminosyren kodet af det kodon.
Forskerne understreger, at resultaterne stadig ikke er i stand til at belyse retningen af årsagssammenhængen, hvilket betyder, at det endnu ikke er muligt at sige, om en ændring i genetisk kodning kan forårsage en ændring i den lokale proteinstruktur, eller om strukturelle ændringer kan forårsage forskellig kodning, for eksempel gennem evolutionære processer. Dette spørgsmål er grundlaget for en efterfølgende forskningsundersøgelse, som gruppen nu udfører. According to Dr. Marx, a biologist by training and education, "If we find in subsequent research that the codon indeed has a causal effect on protein folding, this is likely to have a huge impact on our understanding of protein folding, as well as on future applications, such as engineering new proteins."
Dr. Marx emphasizes that the discovery presented in the article would not have been possible without Prof. Bronstein's computer and analysis skills. "This research is truly interdisciplinary, because biology alone cannot cope with such vast quantities of data without the help of data science, and computer scientists cannot themselves perform research of this kind, since they lack familiarity with the complex biological processes being probed. Therefore, our research highlights the huge advantage of interdisciplinary research that integrates skills from different fields to create a whole that is greater than the sum of its parts."
 Varme artikler
Varme artikler
-
 Hvad betyder inokulering i mikrobiologi?Mikrobiologi er en bestemt gren af videnskabelig undersøgelse, der fokuserer på mikroorganismer. Dette inkluderer bakterier, encellede organismer og ofte vira. Mange udtryk inden for mikrobiologi
Hvad betyder inokulering i mikrobiologi?Mikrobiologi er en bestemt gren af videnskabelig undersøgelse, der fokuserer på mikroorganismer. Dette inkluderer bakterier, encellede organismer og ofte vira. Mange udtryk inden for mikrobiologi -
 Ny infektionssygdom truer Darwins frø med udryddelseDarwins frø vil sandsynligvis blive udslettet af padde svamp. Kredit:University of Zurich Ikoniske arter, der sandsynligvis vil blive udslettet af padde svamp, trods mangel på indlysende kortsigte
Ny infektionssygdom truer Darwins frø med udryddelseDarwins frø vil sandsynligvis blive udslettet af padde svamp. Kredit:University of Zurich Ikoniske arter, der sandsynligvis vil blive udslettet af padde svamp, trods mangel på indlysende kortsigte -
 Masterchef -teknikken viste sig at være en livredder for truede havskildpaddeægKredit:Monash University Forskere fra Monash University har gjort den usandsynlige opdagelse, at en populær Masterchef -teknik kan beskytte æg fra truede havskildpadder under transport. Forskerne
Masterchef -teknikken viste sig at være en livredder for truede havskildpaddeægKredit:Monash University Forskere fra Monash University har gjort den usandsynlige opdagelse, at en populær Masterchef -teknik kan beskytte æg fra truede havskildpadder under transport. Forskerne -
 Udgivet havskildpadde fører oceanografer aske ud på havetSørgende afspejles, når en havskildpadde ved navn Picasso bærer asken fra Tony Amos, 80, en kendt oceanograf, på ryggen, da den frigives tilbage i Den Mexicanske Golf efter en mindehøjtidelighed, Lørd
Udgivet havskildpadde fører oceanografer aske ud på havetSørgende afspejles, når en havskildpadde ved navn Picasso bærer asken fra Tony Amos, 80, en kendt oceanograf, på ryggen, da den frigives tilbage i Den Mexicanske Golf efter en mindehøjtidelighed, Lørd


