AI dechifrerer ny genregulatorisk kode i planter og laver nøjagtige forudsigelser for nyligt sekventerede genomer
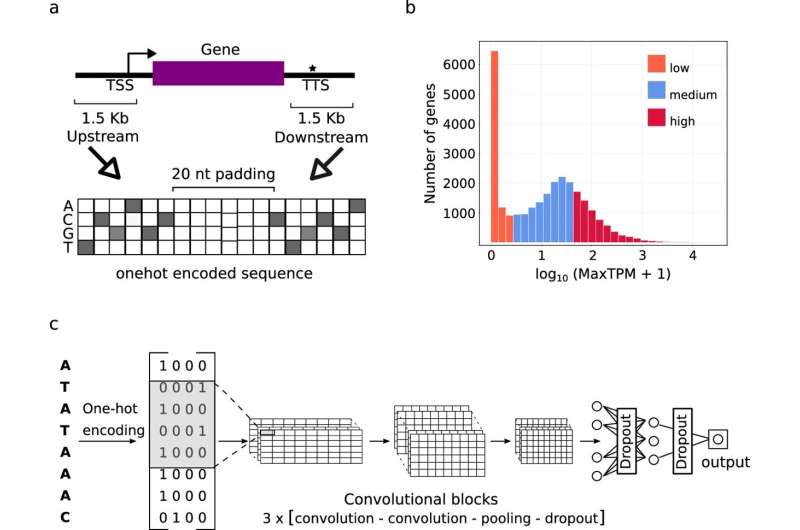
Genom-sekventeringsteknologi giver tusindvis af nye plantegenomer årligt. I landbruget fusionerer forskere denne genomiske information med observationsdata (måling af forskellige planteegenskaber) for at identificere sammenhænge mellem genetiske varianter og afgrødeegenskaber som frøantal, resistens over for svampeinfektioner, frugtfarve eller smag.
Imidlertid er forståelsen af, hvordan genetisk variation påvirker genaktivitet på molekylært niveau, ret begrænset. Denne videnskløft hindrer forædling af "smarte afgrøder" med forbedret kvalitet og reduceret negativ miljøpåvirkning opnået ved kombination af specifikke genvarianter med kendt funktion.
Forskere fra IPK Leibniz Institute og Forschungszentrum Jülich (FZ) har gjort et betydeligt gennembrud for at tackle denne udfordring. Ledet af Dr. Jedrzej Jakub Szymanski trænede det internationale forskerhold fortolkbare deep learning-modeller, en undergruppe af AI-algoritmer, på et stort datasæt af genomisk information fra forskellige plantearter.
"Disse modeller var ikke kun i stand til præcist at forudsige genaktivitet fra sekvenser, men også at finde ud af, hvilke sekvensdele der bidrager til disse forudsigelser," forklarer lederen af IPK's forskningsgruppe "Netværksanalyse og modellering." AI-teknologien, som forskerne anvendte, ligner den, der bruges i computersyn, som involverer genkendelse af ansigtstræk i billeder og udledning af følelser.
I modsætning til tidligere tilgange baseret på statistisk berigelse kombinerede forskerne her identifikation af sekvenstræk med bestemmelse af mRNA-kopitallet i rammen af en matematisk model, der er blevet trænet, der tager højde for biologisk information om genmodelstruktur og sekvenshomologi, således gen evolution.
"Vi var virkelig forbløffede over effektiviteten. Inden for et par dages træning genfandt vi mange kendte regulatoriske sekvenser og fandt ud af, at omkring 50 % af de identificerede funktioner var helt nye. Disse modeller generaliserede fremragende på tværs af plantearter, de ikke blev trænet i, hvilket gjorde dem værdifulde til at analysere nyligt sekventerede genomer," siger Dr. Szymanski.
"Og vi demonstrerede specifikt deres anvendelse i forskellige tomatkultivarer med langlæste sekventeringsdata. Vi udpegede specifikke regulatoriske sekvensvariationer, der forklarede observerede forskelle i genaktivitet og følgelig variationer i form, farve og robusthed. Dette er en bemærkelsesværdig forbedring i forhold til klassisk anvendte statistiske associationer af enkeltnukleotidpolymorfismer."
Holdet har åbent delt deres modeller og leveret en webgrænseflade til deres brug. "Interessant nok gik der en stor indsats i at forringe vores models ydeevne. For at undgå alt for optimistiske resultater på grund af AI, der fandt genveje, krævede jeg et dybt dyk i genreguleringsbiologi for at eliminere enhver potentiel skævhed, reducere datalækage og overtilpasning," siger Fritz Forbang Peleke, den førende maskinlæringsforsker og førsteforfatter af undersøgelsen, som blev offentliggjort i tidsskriftet Nature Communications .
Dr. Simon Zumkeller, en medforfatter og evolutionsbiolog fra FZ Jülich, siger:"Med de præsenterede analyser kan vi undersøge og sammenligne genregulering i planter og udlede dens udvikling. Til praktiske anvendelser giver metoden også et nyt grundlag. Vi nærmer os den rutinemæssige identifikation af genregulerende elementer i kendte og nyligt sekventerede plantegenomer, i forskellige væv og under forskellige miljøforhold."
Flere oplysninger: Fritz Forbang Peleke et al., Deep learning den cis-regulatoriske kode for genekspression i udvalgte modelplanter, Nature Communications (2024). DOI:10.1038/s41467-024-47744-0
Journaloplysninger: Nature Communications
Leveret af Leibniz Institute of Plant Genetics and Crop Plant Research
 Varme artikler
Varme artikler
-
 Mimetoliths:De ansigter, vi ser i klippeformationerThe Old Man of the Mountain levede på et udkanten i Franconia Notch, New Hampshire, og var en af de mest kendte mimetolitter i USA, før den faldt i 2003. Libraray of Congress Et almindeligt begreb,
Mimetoliths:De ansigter, vi ser i klippeformationerThe Old Man of the Mountain levede på et udkanten i Franconia Notch, New Hampshire, og var en af de mest kendte mimetolitter i USA, før den faldt i 2003. Libraray of Congress Et almindeligt begreb, -
 Hvorfor drømmer vi?Få mere søvnbilledgalleri Vi ved ikke, hvorfor vi drømmer, men vi ved, at alle gør det. Se flere søvnbilleder. Bamboo Productions/Getty Images Den menneskelige hjerne er en mystisk lille kugle af gr
Hvorfor drømmer vi?Få mere søvnbilledgalleri Vi ved ikke, hvorfor vi drømmer, men vi ved, at alle gør det. Se flere søvnbilleder. Bamboo Productions/Getty Images Den menneskelige hjerne er en mystisk lille kugle af gr -
 Hvorfor kongebavian-edderkoppegift er så smertefuldtEn King Bavian-edderkop (P. muticus). Kredit:Volker Herzig. Et team af forskere fra Illawarra Health and Medical Research Institute, University of Queenslands Institute for Molecular Bioscience og
Hvorfor kongebavian-edderkoppegift er så smertefuldtEn King Bavian-edderkop (P. muticus). Kredit:Volker Herzig. Et team af forskere fra Illawarra Health and Medical Research Institute, University of Queenslands Institute for Molecular Bioscience og -
 Sådan fungerer pollenPollenkorn tager en endeløs række af fascinerende former med alle slags teksturer og funktioner. Dan Kitwood/Getty Planter udviklede pollen som et reproduktivt middel for mere end 375 millioner år si
Sådan fungerer pollenPollenkorn tager en endeløs række af fascinerende former med alle slags teksturer og funktioner. Dan Kitwood/Getty Planter udviklede pollen som et reproduktivt middel for mere end 375 millioner år si
- Ingeniører fremmer forståelsen af grafeners friktionsegenskaber
- Forskere dechifrerer kulstofs rolle og opdelingen af kontinenter
- Sådan konverteres KBTU til BTU
- Undersøgelse dokumenterer den første menneskelige besættelse i Nordafrika
- Stenpustende bakterier er elektronspindoktorer, undersøgelse viser
- ICESat-2 lasere består den sidste jordprøve


