Ny multi-task dyb læringsramme integrerer storskala enkeltcellet proteomik og transkriptomikdata
De eksponentielle fremskridt inden for enkeltcellede multi-omics-teknologier har ført til akkumulering af store og forskellige multi-omics-datasæt. Integrationen af enkeltcellet proteomik og transkriptomik (eller epigenomik) data udgør imidlertid en betydelig udfordring for eksisterende metoder. Adskillige transformer-baserede modeller, såsom Geneformer, har væsentligt ændret paradigmet for enkeltcellet transkriptomanalyse. Disse metoder stiller imidlertid betydelige krav til beregningsressourcer.
For at imødegå disse udfordringer har forskere ved Wuhan Botanical Garden i det kinesiske videnskabsakademi udviklet en transformer-baseret metode, kaldet scmFormer, til at integrere storskala enkeltcellet proteomik og transkriptomik data ved hjælp af en multi-task transformer. Undersøgelsen med titlen "scmFormer Integrates Large‐Scale Single‐Cell Proteomics and Transcriptomics Data by Multi‐Task Transformer" blev offentliggjort i Advanced Science .
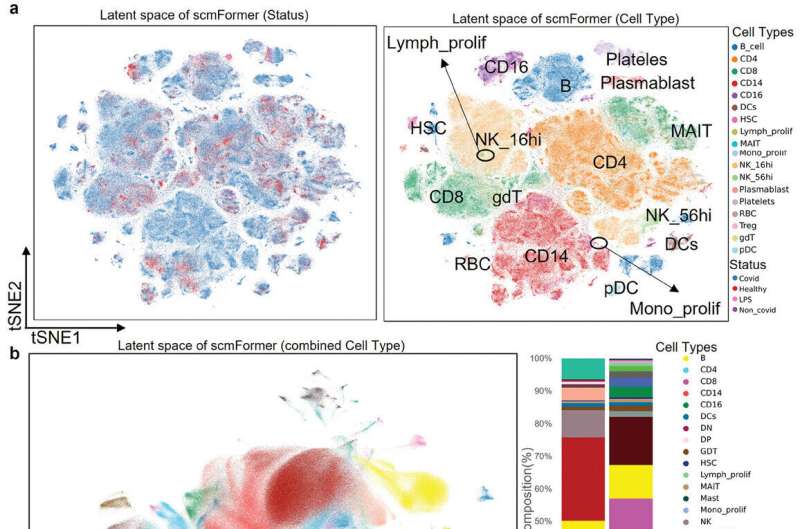
Forskerne præsenterede en omfattende evaluering og lavede casestudier af denne metode, resultaterne viste, at scmFormer udviste bemærkelsesværdig dygtighed til at harmonisere storskala enkeltcellede omics plus proteomics datasæt på både celletype og finere skala celleniveau med begrænsede computerressourcer.
Derudover besidder scmFormer evnen til at integrere flere enkeltcellede parrede multimodale datasæt, hvilket fører til den dobbelte fordel af reducerede høje omkostninger og forbedret biologisk indsigt.
Desuden viser scmFormer en enestående evne til at eliminere tekniske forskelle mellem forskellige omics-modaliteter og samtidig bevare den underliggende biologiske information, der er iboende i dataene, der spænder over både celletyper og eksperimentelle forhold.
Anvendelsen af scmFormer til integration af to COVID-19-datasæt med 1,48 millioner celler demonstrerede yderligere den klare fordel ved scmFormer til håndtering af store datasæt på almindelige bærbare computere.
Flere oplysninger: Jing Xu et al, scmFormer integrerer storskala enkeltcellede proteomik- og transkriptomiske data af Multi-Task Transformer, Advanced Science (2024). DOI:10.1002/advs.202307835
Journaloplysninger: Avanceret videnskab
Leveret af Chinese Academy of Sciences
 Varme artikler
Varme artikler
-
 Ekstra smag og forebyggelse af svindel på menuen for Europas øl- og vinindustriDen europæiske drikkevareindustri vil være foran konkurrenterne ved at forbedre smagen af øl og vin gennem forskning i nye gærstammer. Kredit:Timothy Dykes via Unsplash Europas værdifulde vin- og
Ekstra smag og forebyggelse af svindel på menuen for Europas øl- og vinindustriDen europæiske drikkevareindustri vil være foran konkurrenterne ved at forbedre smagen af øl og vin gennem forskning i nye gærstammer. Kredit:Timothy Dykes via Unsplash Europas værdifulde vin- og -
 Metoden giver forskere mulighed for at indsamle kropslugtprøver af pattedyr på en ikke-invasiv må…Blandt andre muligheder, pattedyr, som surikater, bruge olfaktoriske signaler til at kommunikere med artsfæller. Kredit:Brigitte Schlögl (geb. Weiß) Pattedyr kommunikerer med hinanden ved hjælp af
Metoden giver forskere mulighed for at indsamle kropslugtprøver af pattedyr på en ikke-invasiv må…Blandt andre muligheder, pattedyr, som surikater, bruge olfaktoriske signaler til at kommunikere med artsfæller. Kredit:Brigitte Schlögl (geb. Weiß) Pattedyr kommunikerer med hinanden ved hjælp af -
 Er udryddelse nogensinde en god ting?Skovrydning på steder som Indonesien, afbilledet her, kan forårsage udryddelse og en krusning af negative virkninger i det biologiske samfund. Ulet Ifansasti/Getty Images I begyndelsen af 1950erne,
Er udryddelse nogensinde en god ting?Skovrydning på steder som Indonesien, afbilledet her, kan forårsage udryddelse og en krusning af negative virkninger i det biologiske samfund. Ulet Ifansasti/Getty Images I begyndelsen af 1950erne, -
 Episk hårde baobabtræer, der dør i AfrikaNi af de 13 ældste og fem af de seks største baobabtræforskere, der er undersøgt i løbet af de sidste 12 år, er døde. Rod Waddington/Brugt under Creative Commons CC BY-SA 2.0 Til alle de næsten ubest
Episk hårde baobabtræer, der dør i AfrikaNi af de 13 ældste og fem af de seks største baobabtræforskere, der er undersøgt i løbet af de sidste 12 år, er døde. Rod Waddington/Brugt under Creative Commons CC BY-SA 2.0 Til alle de næsten ubest
- Forskere foreslår ny metode til sporing af undvigende oprindelse af CO₂-emissioner fra vandløb
- Simuleringer tyder på, at en planet på størrelse med Jorden eller Mars lurer ud over Neptun
- Nyt multi-kanals synligt lys-kommunikationssystem bruger en enkelt optisk vej
- Fordele og ulemper ved multidimensionale skalaer
- Minimal tegn på permafrostkulstof i Siberias Kolyma -floden
- Ved begrænsning af kapitalbevægelser, gå ikke alene


