Telescope:et værktøj til at styre bioinformatiske analyser på mobile enheder

Teleskoparkitektur:joblederen indsamler jobinformation ved at oprette forbindelse til målklyngen via sin forbindelsesmanager. Telescopes lokale database opbevarer disse oplysninger, som gengives af brugergrænsefladen til en (mobilvenlig) webside. Kredit:Brito et al.
Et team af forskere ved UCLA, universitetet i São Paulo, Federal University of São Carlos og University of Southern California har for nylig udviklet et interaktivt værktøj til styring af bioinformatiske analyser i stor skala i realtid og fra bærbare enheder. Dette nye værktøj, kaldet Teleskop, blev første gang præsenteret i et papir, der var forudgivet den arXiv .
Holdet, der udviklede Telescope, omfatter postdoc-stipendiater fra UCLA Collaboratory, et forskningslaboratorium ved universitetets institut for kvantitative og beregningsmæssige biovidenskaber. At arbejde på UCLA Collaboratory giver disse forskere mulighed for at interagere med andre studerende og fakultetsmedlemmer på universitetet, som har forskellige niveauer af ekspertise inden for beregningsbiologi. Disse interaktioner er det, der i sidste ende inspirerede teamet til at udvikle et brugervenligt værktøj til styring af store bioinformatiske analyser.
"De fleste biovidenskabelige og biomedicinske undersøgelser i dag retter sig mod effekter så subtile og svære at opdage, at store mængder data og beregningskraft er blevet den nye status quo, " fortalte forskerne TechXplore via e-mail. "Ofte, imidlertid, forskere beskriver den overdrevne afhængighed af kommandolinjeværktøjer som besværlig og meget uintuitiv for dem, der mangler formel beregningsuddannelse."
Specialiserede uddannelsesinitiativer og workshops har vist sig at være særligt effektive til at tilskynde forskere til at begynde at bruge kraftfulde metoder til storskala bioinformatiske analyser. Imidlertid, de fleste teknikker til at få adgang til disse metoder på beregningsfaciliteter er ikke særlig godt integreret med våde laboratoriemiljøer, hvor de fleste biologiske forsøg udføres. Med det i tankerne, forskerne satte sig for at udvikle et værktøj, der ville muliggøre bedre integration mellem bioinformatikværktøjer og vådt laboratorieforskning.
"Teleskop blev foreslået for at fremme integration og tættere samarbejde mellem vådt laboratorieforskning og bioinformatik, reduktion af den indlæringskurve, der traditionelt kræves for at køre komplekse bioinformatiske analyser i beregningsfaciliteter, " fortalte teamet TechXplore. "Udfordringen, imidlertid, garanterede, at den endelige løsning faktisk ville løse hullerne og være intuitiv for de fleste forskere."
For at præsentere deres løsning for både teknologikyndige forskere og dem med begrænsede computerfærdigheder, forskerne besluttede at introducere Telescope under II Hackathon-arrangementet i begyndelsen af 2018. Hackathon samler forskere med IT- og kodningsfærdigheder, give dem mulighed for at lære it-færdigheder som en gruppe, for eksempel, udforske, hvordan man bygger scripts og anvender bioinformatikværktøjer til datasæt.
"Vores idé tiltrak både nybegyndere, der ønskede at få deres hænder til at arbejde direkte med data så hurtigt som muligt, og teknologikyndige, der bare ville have en nemmere måde at tjekke deres arbejde på fra deres telefoner, "Forskerne forklarede. "Teleskopet blev således designet fra og for samfundet."
De fleste forskere, der udfører eksperimenter i våde laboratoriemiljøer, skal opfylde betydelige tidskrav. Telescope er designet til at hjælpe dem med at styre deres arbejdsbyrde bedre, supervision og justering af bioinformatikanalyser i realtid.
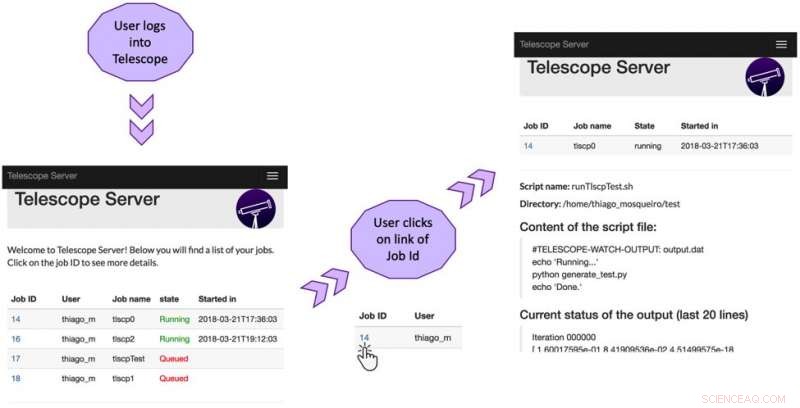
Teleskopbrugergrænseflade:Den første skærm viser status for jobs på klyngen. Det næste skærmbillede viser detaljerede oplysninger om det første job på listen:kildekatalog, navn og indhold af scriptfilen, og sidste linjer af aktuel opgaveoutput. Kredit:Brito et al.
I det væsentlige, Telescope styrker forholdet mellem våde laboratorieeksperimenter og værktøjer til at analysere store mængder biologiske data. Det gør det ved at tilbyde en sikker og brugervenlig platform, hvor brugerne kan kickstarte deres forskning og administrere opgaver, der er relevante for bioinformatikjobs, der kører i højtydende klynger.
"Telescope udnytter almindelige web-frameworks såsom Twitters Bootstrap til at give en eye-candy brugeroplevelse og understøtte en udvidelsesbar grænseflade, " sagde forskerne. "At hensigt at støtte både nye og erfarne brugere, Telescopes kerne administrerer og gemmer brugeradgangsnøgler ved hjælp af industristandarder."
Adgangsnøglerne gemt af Telescope bruges til at forbinde højtydende beregningsklynger gennem en sikker shell for at modtage de seneste jobstatusopdateringer og udstede nye kommandoer, når det er nødvendigt (f.eks. sletning af gamle opgaver osv.). Telescope giver vådlab-forskere mulighed for at spore status for deres arbejde og nemt få adgang til foreløbige resultater af deres bioinformatikanalyser direkte på deres mobiltelefoner. Det betyder, at de også tidligt kan identificere potentielle problemer med deres analyser og annullere dem uden at skulle bruge en computer.
"Sammenlignet med den sædvanlige tilgang til at bruge kommandolinjer på Linux-terminaler, Telescope er et mere intuitivt og interaktivt værktøj, " forklarede forskerne. "F.eks. at dele analyseresultater er lige så nemt som at dele et link, som du ville gøre på Twitter eller Facebook. Ikke desto mindre, vi føler, at vores mest meningsfulde præstation var engagementet med det traditionelt ikke-beregningsmæssige life science-samfund i begyndelsen af udviklingen."
Dette nye værktøj til styring af bioinformatiske analyser tog nøje hensyn til biologiforskernes behov. Faktisk, holdet benyttede sig af Hackathon-begivenheden til at indsamle input og feedback fra biologieksperter med forskellige grader af it-kompetence og erfaring med brug af beregningsværktøjer.
"Vi indsamlede input fra uerfarne computerbrugere, da vi ønskede at skabe et værktøj, der tackler de mest udfordrende punkter ved at bruge uintuitive kommandolinjer, " sagde forskerne. "Vi udviklede derefter en enkel og minimal webgrænseflade, der kan køre på forskellige enheder, herunder mobiltelefoner, med nul opsætning. Udfordringen var at afveje enkelhed med sikkerhed, og vi opnåede en god balance ved at anvende industristandardprotokoller, såsom lagring af nøgler ved hjælp af PBDKS."
Værktøjet udviklet af denne gruppe forskere, som nu er tilgængelig på GitHub, kan hjælpe biologieksperter i deres arbejde, hjælpe dem med at administrere deres bioinformatiske analyser på en enklere og mere intuitiv måde. Ud over at give biomedicinske forskere adgang til kraften i store beregningsfaciliteter fra deres bærbare enheder, Telescope er en software, der kan tilpasses og udvides. Det betyder, at brugerne kan tilføje nye funktioner til den eller være med til at udvikle den yderligere.
"Vores projekt vil nu blive vedligeholdt af Mangul Lab ved USC som open source-software, " sagde forskerne. "Vi planlægger også at integrere Telescope i de fleste af vores laboratoriums analysepipelines for at evaluere brugeroplevelsen yderligere. Som næste skridt, disse tests vil blive udvidet til en meningsmåling af betatestere fra et bredere fællesskab. Vores overordnede mål er at udrulle Telescope for brugerne af USC's højtydende klynge i fremtiden. Gennem hele denne proces, Mangul-laboratoriet vil trives med at engagere sig i samfundet for at fremme adoption og sikre, at Telescope forbliver relevant."
© 2019 Science X Network
 Varme artikler
Varme artikler
-
 Ikke-giftig prototype af saltvandsbatteri kan oplades på få sekunderKredit:Imperial College London En batteriprototype er designet med saltvand og materialer, der er giftfri og oplades hurtigt, baner vejen for nye batterityper. Designprincipperne bag den nye prot
Ikke-giftig prototype af saltvandsbatteri kan oplades på få sekunderKredit:Imperial College London En batteriprototype er designet med saltvand og materialer, der er giftfri og oplades hurtigt, baner vejen for nye batterityper. Designprincipperne bag den nye prot -
 Fiat Chrysler åbner ny fabrik i Detroit:rapportJeep Grand Cherokee vil rulle ud af produktionslinjen i Detroit på en ny Fiat Chrysler-fabrik på grund af åbning, det første nye bilsamlebånd, der åbnede i Motor City i 27 år Fiat Chrysler åbner e
Fiat Chrysler åbner ny fabrik i Detroit:rapportJeep Grand Cherokee vil rulle ud af produktionslinjen i Detroit på en ny Fiat Chrysler-fabrik på grund af åbning, det første nye bilsamlebånd, der åbnede i Motor City i 27 år Fiat Chrysler åbner e -
 Petrobras -overskuddet springer i andet kvartal på grund af højere oliepriserResultaterne tyder på, at Petrobras vender om efter et kraftigt fald i råpriserne og en massiv korruptionsskandale i virksomheden Brasiliens flagskibsolieselskab Petrobras annoncerede kraftigt høj
Petrobras -overskuddet springer i andet kvartal på grund af højere oliepriserResultaterne tyder på, at Petrobras vender om efter et kraftigt fald i råpriserne og en massiv korruptionsskandale i virksomheden Brasiliens flagskibsolieselskab Petrobras annoncerede kraftigt høj -
 Luftfartspiloter kæmper med at fortolke vejrudsigter og observationsskærmeI april 2018-udgaven af International Journal of Aerospace Psychology, Embry-Riddles Elizabeth Blickensderfer, en professor ved Institut for Menneskelige Faktorer og Behavioral Neurobiologi, rapport
Luftfartspiloter kæmper med at fortolke vejrudsigter og observationsskærmeI april 2018-udgaven af International Journal of Aerospace Psychology, Embry-Riddles Elizabeth Blickensderfer, en professor ved Institut for Menneskelige Faktorer og Behavioral Neurobiologi, rapport
- Globale flyselskaber skærer næsten alle flyvninger ned, da virus spreder vinger
- Afdækning af mysteriet om betydningen af Påskeøerne Moai
- Cold Desert Plants and Animals
- Debunking undersøgelse foreslår måder at imødegå misinformation og rette falske nyheder
- Forholdsregler under cykloner
- Cleveland Abbe


