Beregningsmetode giver hurtigere højopløsnings massespektrometrisk billeddannelse
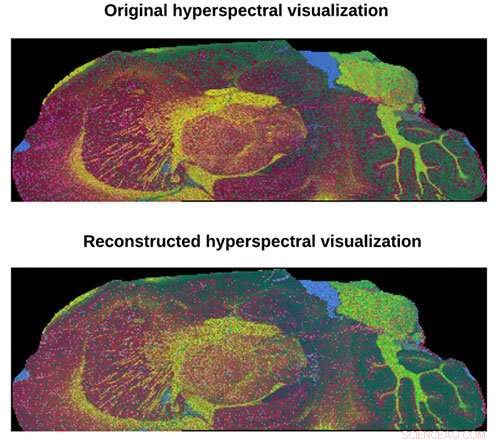
Top, hyperspektral visualisering med data fra et standard 9-timers eksperiment sammenlignet med hyperspektral visualisering med data fra et foreslået 1-timers eksperiment. Kredit:Beckman Institute for Advanced Science and Technology
En ny beregningsmæssig massespektrometrisk billeddannelsesmetode gør det muligt for forskere at opnå høj masseopløsning og høj rumlig opløsning for biologiske prøver, samtidig med at de leverer datasæt eksponentielt hurtigere.
Forskere ved Beckman Instituttet for Avanceret Videnskab og Teknologi udviklede en subspace massespektrometri-billedmetode, der accelererer dataindsamlingshastigheden - uden at ofre kvaliteten - ved at designe en modelbaseret rekonstruktionsstrategi.
Teknikken, som blev udviklet ved hjælp af dyremodeller, kan have vigtige konsekvenser for mange applikationer, herunder analytisk kemi og kliniske undersøgelser, med resultater tilgængelige på en brøkdel af tiden. Det kan også detektere en lang række biomolekyler - fra små molekyler såsom neurotransmittere og aminosyrer til større molekyler såsom lipider eller peptider.
Artiklen "Accelerating Fourier Transform-Ion Cyclotron Resonance Mass Spectrometry Imaging Using a Subspace Approach" blev offentliggjort i Journal of American Society of Mass Spectrometry .
"Fourier transformation-ion cyklotronresonans er et virkelig kraftfuldt instrument, giver den højeste masseopløsning, " sagde Yuxuan Richard Xie, en bioingeniørstuderende ved University of Illinois Urbana-Champaign, hvem er første forfatter på papiret. "Men en ulempe ved FT-ICR er, at den er meget langsom. Så i bund og grund, hvis folk ønsker at opnå en vis masseopløsning, de skal vente dage på at få datasæt. Vores beregningsmæssige tilgang fremskynder denne opkøbsproces, potentielt fra en dag til måske en til to timer – dybest set en tidobling af dataindsamlingshastigheden."
"Vores metode ændrer den måde, vi indhenter data på, " sagde Xie. "I stedet for at erhverve massespektre pr. pixel, Teknikken genkender redundansen i de højdimensionelle billeddata og bruger en lavdimensionel underrumsmodel til at udnytte denne redundans til at rekonstruere multispektrale billeder fra kun en delmængde af dataene."
Xie samarbejdede med Fan Lam, en adjunkt i bioteknik, og Jonathan V. Sweedler, James R. Eiszner Family Endowed Chair i Kemi og direktøren for School of Chemical Sciences, som er co-principal investigators på papiret. Daniel Castro, en kandidatstuderende i molekylær og integrativ fysiologi, også bidraget.
"Vi har brugt subspace-modeller i vores MR- og MR-spektroskopiske billeddannelsesarbejde i lang tid, " sagde Lam. "Det er virkelig rart at se, at det også har store potentialer for en anden biokemisk billeddannelsesmodalitet."
"Evnen til at tilegne sig forbedret kemisk information og placeringen af kemikalierne i en kompleks prøve, såsom en del af en hjerne, gør det muligt for vores neurokemiske forskning, " sagde Sweedler.
Underrums billedbehandlingskonceptet blev udviklet af Zhi-Pei Liang, en professor i elektro- og computerteknik og fuldtidsansatte Beckman-fakultet, som er en verdensførende ekspert i MR og MRSI.
Forskningen fortsætter, mens forskere søger at anvende teknikken til 3-D-billeddannelse. "(Tilgangen) kunne have en meget større indflydelse for det videnskabelige samfund for 3-D-billeddannelse af større områder, såsom hjernen, " sagde Xie. "For hvis vi laver 50 skiver på FT-ICR, det ville tage uger lige nu, men (med denne teknik) kan vi opnå en anstændig dækning måske inden for få dage.
"Jeg tror på, at databehandling, især den datadrevne tilgang, er som en ny lysende stjerne. Det bliver mere og mere kraftfuldt, og vi bør bestemt bruge nogle af disse teknikker til kemisk analyse af væv gennem massespektrometrisk billeddannelse."
Sidste artikelNår kemi med grønt lys efterligner, hvad der sker i livet
Næste artikelHvordan skadelige proteiner dannes
 Varme artikler
Varme artikler
-
 Forskning i, hvordan man skaber selvrensende overflader via 3-D-printJuan Manuel Barrios og Pablo Romero, undersøgelsens efterforskere. Kredit:Universidad de Córdoba (Spanien) Det bliver mere og mere indlysende, at 3-D-print er mere end blot et interessant legetøj,
Forskning i, hvordan man skaber selvrensende overflader via 3-D-printJuan Manuel Barrios og Pablo Romero, undersøgelsens efterforskere. Kredit:Universidad de Córdoba (Spanien) Det bliver mere og mere indlysende, at 3-D-print er mere end blot et interessant legetøj, -
 Chemist foreslår ny metode til at syntetisere prækursorer til Parkinsons-lægemidlerKredit:RUDN Universitet En kemiker fra RUDN University har foreslået en ny metode til syntese af sekundær propargylamin, der bruges til at skabe antidepressiva, medicin mod Parkinsons sygdom og an
Chemist foreslår ny metode til at syntetisere prækursorer til Parkinsons-lægemidlerKredit:RUDN Universitet En kemiker fra RUDN University har foreslået en ny metode til syntese af sekundær propargylamin, der bruges til at skabe antidepressiva, medicin mod Parkinsons sygdom og an -
 Mikrober kunne gøre medicinproduktionen mere effektivAlkaloidbaserede lægemidler afledt af planter kan være potente behandlinger for en række forskellige sygdomme. Men at få disse kraftfulde terapeutiske midler fra planter kan tage lang tid og koste man
Mikrober kunne gøre medicinproduktionen mere effektivAlkaloidbaserede lægemidler afledt af planter kan være potente behandlinger for en række forskellige sygdomme. Men at få disse kraftfulde terapeutiske midler fra planter kan tage lang tid og koste man -
 Forskere udvikler værktøj til at hjælpe med udvikling, effektiviteten af brintdrevne bilerSkematisk indre funktion af elektroderne i en brændselscelle, og vigtigheden af nøgleparametre. Kredit:Heinz et al., 2021 Udbredt anvendelse af brintdrevne køretøjer frem for traditionelle elekt
Forskere udvikler værktøj til at hjælpe med udvikling, effektiviteten af brintdrevne bilerSkematisk indre funktion af elektroderne i en brændselscelle, og vigtigheden af nøgleparametre. Kredit:Heinz et al., 2021 Udbredt anvendelse af brintdrevne køretøjer frem for traditionelle elekt
- Indien lider under det varmeste årti nogensinde
- De mest effektive individuelle trin til at tackle klimaforandringer diskuteres ikke
- Klimatilpasning kræver milliarder i ekstra årlige investeringer for at forhindre sult
- Metamaterialer åbner helt nye muligheder inden for optik
- Intensiv biomassehøst forbundet med brandmyrekolonisering, nedsat mangfoldighed af hvirvelløse dyr
- Ikke-fugle dinosaurer fundet at have lagt blå æg


