Kyllingeembryoner giver værdifulde genetiske data til at forstå menneskelig udvikling
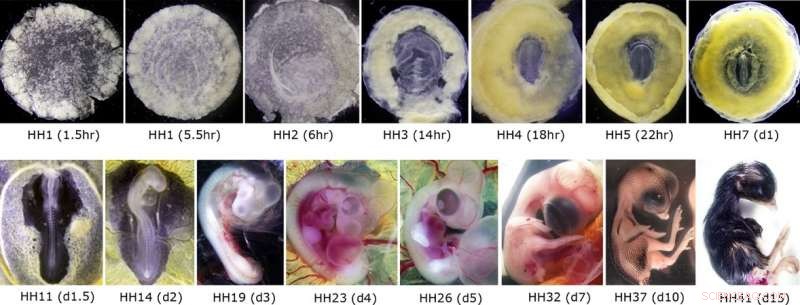
Repræsentative billeder af kyllingeembryoner brugt til RNA-forberedelse og efterfølgende CAGE-sekventering. Øverste paneler viser embryoner på den første dag af embryonal udvikling (21 dage i alt), svarende til de første fem uger af menneskelig svangerskab (første tre uger af menneskelig udvikling efter befrugtning). Nederste paneler viser embryoner fra etableringen af kredsløbssystemet til omkring en uge før klækning, svarende til begyndelsen af tredje trimester i menneskelig udvikling. Kredit:Professor Guojun Sheng
Tidlig udviklingsforskning på menneskelige embryoner er umulig på grund af etiske overvejelser, og er teknisk vanskelig at udføre hos andre pattedyr, gør ressourcer til denne type forskning yderst sjældne. For eksempel, Functional Annotation of Mammalian Genome (FANTOM) 5-projekt baseret i Japan, etableret i 2000, er et verdensomspændende samarbejdsprojekt med det mål at identificere alle funktionelle elementer i pattedyrs genomer, men det har ingen Cap Analysis of Gene Expression (CAGE)-baseret transkriptionsstartsted (TSS) data for humane embryoner eller for murine embryoner, der repræsenterer tidlige udviklingsstadier.
For at komme uden om denne mangel på information, et internationalt samarbejde mellem forskere fra Japan (Kumamoto University &RIKEN), Rusland (Kazan Federal University), Spanien (University of Cantabria), og Australien (University of Western Australia) har sammensat verdens første genom-dækkende sæt af fugle-TSS'er. Disse steder markerer udgangspunktet for transkription og er ekstremt vigtige for celledifferentiering og embryoudvikling indtil fødslen, eller, i tilfælde af en fugleart, indtil udklækning. Mens fugle ser ganske anderledes ud end mennesker, tidlige udviklingsstadier er meget ens, og det er blevet vist, at omkring 60 % af de humane proteinkodende gener har en-til-en-korrespondance med kyllingeortologer (gener, der er udviklet fra en fælles forfader).
Ved at bruge CAGE-teknologi, en yderst pålidelig metode til at finde TSS'er og cis-regulatoriske elementer (de sekvensblokke, der regulerer positionen og robustheden af transkription) i genomet over hele kyllingens udviklingsperiode, samarbejdspartnerne var i stand til at kortlægge 60 % af alle udviklingsmæssige kyllinge-TSS'er til det seneste kyllingegenom, med de øvrige 40 % sandsynligvis endnu ikke-karakteriserede alternative promotorer eller ikke-kodende RNA-gener. Det opdaterede genom blev tilføjet til RIKENs interaktive, online platform ZENBU (det japanske ord "zenbu" oversættes til "alle" på engelsk.) under navnet "Chicken-ZENBU" og er gratis for alle at bruge.
Forskerne viste også, at deres genomomfattende TSS-kortlægning kunne anvendes til CRISPR-on-teknologi for at aktivere specifikke gener under udvikling ved succesfuldt at aktivere T-genet for Brachyury-protein, der koder på udviklingsstadiet HH10 (ca. 1,5 dages udvikling efter lægning). Denne præstation blev fulgt op af flere mere vellykkede genaktiveringer, hvilket viste effektiviteten af CRISPR-on, når det kombineres med TSS-kortlægning.
"I processen med at udvikle vores CAGE-baserede udviklingsmæssige TSS-profileringsmetode, vi identificerede nye transkriptionsfaktorer og deres regulatoriske moduler, samt flere nye husholdningsgener, " sagde projektleder professor Guojun Sheng fra Kumamoto University's IRCMS. "Vi forventer, at dette arbejde og de data, vi føjede til ZENBU online-databasen, vil i høj grad fremme udviklingsforskningen i både fugle og pattedyr."
 Varme artikler
Varme artikler
-
 Cyanobakterielle undersøgelser undersøger cellulær struktur under nitrogensultForskere fra Washington University i St. Louis og ORNL bruger neutroner til at studere, hvad der sker, når celleprøver af cyanobakterier (billedet) sultes efter nitrogen. De er især interesserede i, h
Cyanobakterielle undersøgelser undersøger cellulær struktur under nitrogensultForskere fra Washington University i St. Louis og ORNL bruger neutroner til at studere, hvad der sker, når celleprøver af cyanobakterier (billedet) sultes efter nitrogen. De er især interesserede i, h -
 At få myg til at destruere sig selvCRISPR/Cas9-medieret forstyrrelse af gener forbundet med øjenpigment fik øjenfarven til at ændre sig fra sort til hvid. Kredit:Michelle Bui, UC Riverside. Forskere ved University of California, Ri
At få myg til at destruere sig selvCRISPR/Cas9-medieret forstyrrelse af gener forbundet med øjenpigment fik øjenfarven til at ændre sig fra sort til hvid. Kredit:Michelle Bui, UC Riverside. Forskere ved University of California, Ri -
 Risiko for kejsersnit er arvelig - Naturlig selektion kan ikke reducere antallet af forhindret arbej…Biolog Philipp Mitteroecker fandt ud af, at kvinder født af kejsersnit på grund af en fetopelvisk disproportion (FDP) er mere end dobbelt så tilbøjelige til at udvikle FDP, når de føder, end kvinder f
Risiko for kejsersnit er arvelig - Naturlig selektion kan ikke reducere antallet af forhindret arbej…Biolog Philipp Mitteroecker fandt ud af, at kvinder født af kejsersnit på grund af en fetopelvisk disproportion (FDP) er mere end dobbelt så tilbøjelige til at udvikle FDP, når de føder, end kvinder f -
 Amylase-aktivitet i mavenTalrige enzymer er involveret i nedbrydning af forskellige forbindelser i fødevarer, når det passerer gennem fordøjelseskanalen. Amylase findes i to hovedområder - spyt i munden og bugspytkirtelsaf
Amylase-aktivitet i mavenTalrige enzymer er involveret i nedbrydning af forskellige forbindelser i fødevarer, når det passerer gennem fordøjelseskanalen. Amylase findes i to hovedområder - spyt i munden og bugspytkirtelsaf
- Stonehenge var udlændinges sidste hvilested,
- Fireball-meteorit indeholder uberørte udenjordiske organiske forbindelser
- Koldtvandsstrømme kan give tilflugtssted mod skiftende klima
- Den komplicerede fremtid for havvindkraft i USA
- Hacking af den menneskelige hjerne - laboratoriefremstillede synapser til kunstig intelligens
- Mindre og skarpere:Nanoscale Metal-Organic Frameworks (NMOF'er) som MRI-kontrastmidler


