Genteknisk mekanisme visualiseret
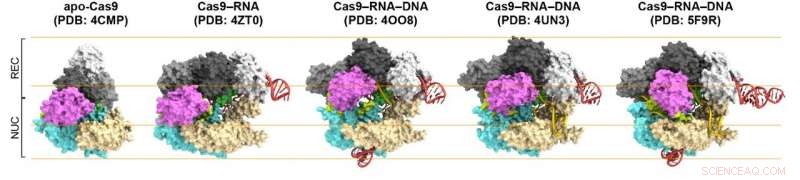
Figur 1. Strukturer af Cas9. Fra venstre til højre:Cas9 alene (apo-Cas9), Cas9 bundet til RNA (Cas9 – RNA), Cas9 – RNA bundet til sit enkeltstrengede DNA-mål (Cas9 – RNA – DNA), Cas9 – RNA bundet til en delvis DNA-duplex (Cas9 – RNA – DNA) og Cas9 – RNA bundet til sit dobbeltstrengede DNA-mål (Cas9 – RNA – DNA). Kredit:Kanazawa University
Forskere ved Kanazawa University og University of Tokyo rapporterer i Naturkommunikation visualisering af dynamikken i 'molekylær saks'-hovedmekanismen i genteknologisk teknik CRISPR-Cas9.
En af de teknikker, der anvendes inden for genteknologi-processen med kunstig ændring af genomet for en levende organisme-involverer det såkaldte CRISPR-Cas9-nukleasesystem. Ved hjælp af dette system, en celles DNA kan skæres på et ønsket sted, hvor gener kan slettes eller tilføjes. Valg af stedet, der skal skæres, foretages ved hjælp af et 'guide RNA' -molekyle bundet til Cas9 -proteinet. Nu, et team af forskere ledet af Mikihiro Shibata fra Kanazawa University og Osamu Nureki fra University of Tokyo har visualiseret dynamikken i CRISPR-Cas9-komplekset, især hvordan det skærer DNA, giver værdifuld indsigt i den CRISPR-Cas9-medierede DNA-spaltningsmekanisme.
Til deres visualiseringsstudier, forskerne brugte højhastigheds atomkraftmikroskopi (HS-AFM), en metode til billeddannelse af overflader. En overflade sonderes ved at flytte en lille cantilever hen over den; kraften, som sonden oplever, kan omdannes til et højdemål. En scanning af hele overfladen resulterer derefter i et højdekort over prøven. Den hurtige eksperimentelle opsætning af Shibata og kolleger muliggjorde ekstremt hurtig, gentagne scanninger - konverterbare til film - af biomolekylerne, der deltager i den molekylære sakseaktion.
Først, forskerne sammenlignede Cas9 uden og med RNA vedhæftet (Cas9 – RNA). De fandt ud af, at førstnævnte var i stand til fleksibelt at vedtage forskellige konformationer, mens sidstnævnte har en fast, to-lap struktur, fremhæver konformationsstabiliseringsevnen af guide-RNA'et. Derefter, Shibata og kolleger kiggede på, hvordan det stabiliserede Cas9 -RNA -kompleks retter sig mod DNA. De bekræftede, at det binder sig til et på forhånd udvalgt protospacer-tilstødende motiv (PAM) sted i DNA'et. En PAM er en kort nukleotidsekvens placeret ved siden af DNA'ets målsted, som er komplementær til guide -RNA.
Forskerteamets højhastighedsfilm afslørede yderligere, at målretning ('DNA-forhør') opnås gennem 3-D-diffusion af Cas9-RNA-komplekset. Endelig, det lykkedes forskerne at visualisere dynamikken i selve spaltningsprocessen:de observerede, hvordan regionen 'molekylær saks' undergår konformationsudsving, efter at Cas9 – RNA lokalt afvikler det dobbeltstrengede DNA (film 1 [URL]).
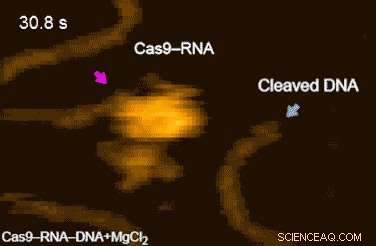
Figur 2. HS-AFM film af DNA-spaltning med Cas9-RNA. Udsving i nukleasedomænet er angivet med magentapile. Spaltningsprodukter frigivet fra Cas9 – RNA er angivet med blå pile. Kredit:Kanazawa University
Shibatas arbejde fremmer vores forståelse af genomredigeringsmekanismen CRISPR-Cas9. Med forskernes ord:"... denne undersøgelse giver hidtil usete detaljer om den funktionelle dynamik i CRISPR-Cas9, og fremhæver HS-AFM's potentiale for at belyse virkningsmekanismerne for RNA-styrede effektor-nukleaser fra forskellige CRISPR-Cas-systemer. "
CRISPR-Cas9
CRISPR, kort for "klyngede regelmæssigt mellemrum korte palindromiske gentagelser", refererer til et sæt bakterielle DNA -sekvenser, der indeholder fragmenter af DNA'et fra vira, der tidligere har angrebet bakterierne. Disse fragmenter bruges af bakterierne til at forhindre yderligere angreb af de samme vira. "Cas" refererer til CRISPR-associerede gener; "Cas9" er et CRISPR-associeret protein med to nukleasedomæner (En nuklease er et enzym, der er i stand til at spalte nukleinsyrer, organiske molekyler til stede i DNA og RNA).
I de seneste år, en genteknisk teknik, hvor et CRISPR-Cas9-kompleks fungerer som 'molekylær saks' er blevet udviklet Cas9 -nukleasen binder til et guide -RNA -molekyle, der indeholder information om DNA -stedet, der skal målrettes. Ved hjælp af højhastigheds atomkraftmikroskopi, Mikihiro Shibata fra Kanazawa University og kolleger har nu undersøgt dynamikken i CRISPR-Cas9-komplekset i detaljer.
Atomkraftmikroskopi
Atomic force microscopy (AFM) er en billeddannelsesteknik, hvor billedet dannes ved at scanne en overflade med en meget lille spids. Horizontal scanning motion of the tip is controlled via piezoelectric elements, while vertical motion is converted into a height profile, resulting in a height distribution of the sample's surface. As the technique does not involve lenses, its resolution is not restricted by the so-called diffraction limit. In a high-speed setup, AFM can be used to produce movies of a sample's evolution in real time. High-speed AFM has been used successfully to study protein dynamics, for example myosin V walking on an actin filament, the photo-induced conformational change of bacteriorhodopsin, and the degradation of cellulose. Shibata and colleagues have now applied the high-speed AFM technique for visualizing the dynamics of DNA cleavage by CRISPR-Cas9.
 Varme artikler
Varme artikler
-
 En ingeniørguide til embryonetXenopus haletudser er fremragende testpersoner, fordi deres gennemsigtige kroppe giver uhindret udsigt til deres indre anatomi. Kredit:MechMorpho Lab/Lance Davidson Om cirka 48 timer, den enkelte
En ingeniørguide til embryonetXenopus haletudser er fremragende testpersoner, fordi deres gennemsigtige kroppe giver uhindret udsigt til deres indre anatomi. Kredit:MechMorpho Lab/Lance Davidson Om cirka 48 timer, den enkelte -
 Kapløb for at redde indonesisk krokodille, der er ramt af dækhalskædeNaturfredningsfolk mener, at nogen bevidst kan have placeret dækket rundt om det beskyttede dyrs hals i et mislykket forsøg på at fange det som et kæledyr Indonesiske bevaringsmyndigheder er i ræs
Kapløb for at redde indonesisk krokodille, der er ramt af dækhalskædeNaturfredningsfolk mener, at nogen bevidst kan have placeret dækket rundt om det beskyttede dyrs hals i et mislykket forsøg på at fange det som et kæledyr Indonesiske bevaringsmyndigheder er i ræs -
 Trods skovtab, et afrikansk beskyttet område har stadig potentiale til at støtte titusindvis af el…Der er meget få steder tilbage på planeten, som kan rumme bestande af store dyreliv i titusindvis, og Niassa Reserve, med dens forbindelse til Selous Game Reserve i det sydlige Tanzania, er et af diss
Trods skovtab, et afrikansk beskyttet område har stadig potentiale til at støtte titusindvis af el…Der er meget få steder tilbage på planeten, som kan rumme bestande af store dyreliv i titusindvis, og Niassa Reserve, med dens forbindelse til Selous Game Reserve i det sydlige Tanzania, er et af diss -
 Forskere identificerer skjult genetisk variation, der hjælper med at drive evolutionenFørsteforfatter Mahul Chakraborty ser gennem flere eksemplarer af frugtfluer for at identificere nye fænotyper. Kredit:UCI Det har været vanskeligt at identificere komplekse mutationer i strukture
Forskere identificerer skjult genetisk variation, der hjælper med at drive evolutionenFørsteforfatter Mahul Chakraborty ser gennem flere eksemplarer af frugtfluer for at identificere nye fænotyper. Kredit:UCI Det har været vanskeligt at identificere komplekse mutationer i strukture
- Ansigt til ansigt astronomikonference bidrager med 3, 000 gange mere kuldioxid end onlinekonference
- Forskning i ren luft omdanner giftig luftforurening til industrikemikalie
- Bangladesh lukker mobilt internet ned for at tackle teenageprotester
- dødstrusler, trolling almindeligt for forskere, der taler til medier om COVID
- Hvordan elektronisk hud kunne hjælpe mennesker med handicap
- AT&T trækker Friends fra Netflix til sin streamingtjeneste


