Revolutionær billedbehandlingsteknik bruger CRISPR til at kortlægge DNA-mutationer
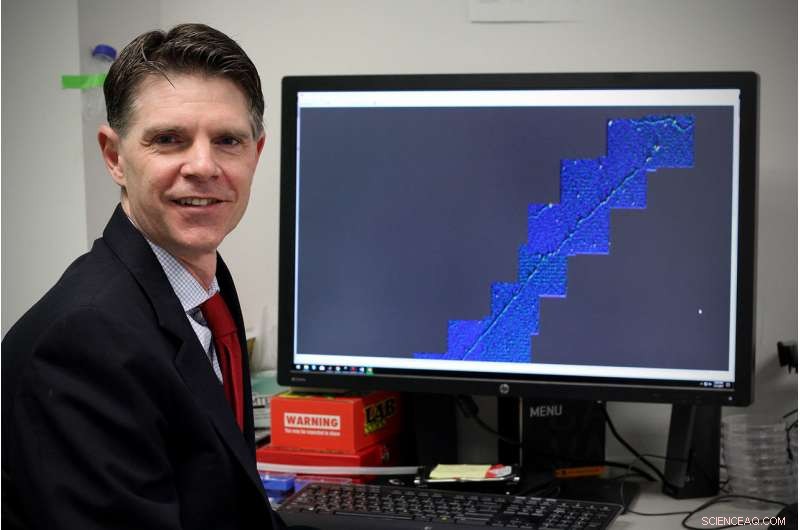
Jason Reed, Ph.D., medlem af Cancer Molecular Genetics forskningsprogrammet ved VCU Massey Cancer Center og lektor i Institut for Fysik ved VCU College of Humanities and Sciences. Kredit:John Wallace ved VCU Massey Cancer Center
Et team af videnskabsmænd ledet af Virginia Commonwealth University fysiker Jason Reed, Ph.D., har udviklet ny nanomapping-teknologi, der kan ændre måden, hvorpå sygdomsfremkaldende genetiske mutationer diagnosticeres og opdages. Beskrevet i en undersøgelse offentliggjort i dag i tidsskriftet Naturkommunikation , denne nye tilgang anvender højhastigheds-atomkraftmikroskopi (AFM) kombineret med en CRISPR-baseret kemisk stregkodningsteknik til at kortlægge DNA næsten lige så præcist som DNA-sekventering, mens store dele af genomet behandles med en meget hurtigere hastighed. Hvad mere er – teknologien kan drives af dele, der findes i din banebrydende DVD-afspiller.
Det menneskelige genom består af milliarder af DNA-basepar. Optrævlet, den strækker sig til en længde på næsten seks fod lang. Når celler deler sig, de skal lave en kopi af deres DNA til den nye celle. Imidlertid, nogle gange kopieres forskellige dele af DNA'et forkert eller klistres sammen på det forkerte sted, fører til genetiske mutationer, der forårsager sygdomme som kræft. DNA -sekventering er så præcis, at den kan analysere individuelle basepar af DNA. Men for at analysere store dele af genomet for at finde genetiske mutationer, teknikere skal bestemme millioner af bittesmå sekvenser og derefter sætte dem sammen med computersoftware. I modsætning, biomedicinske billeddannelsesteknikker såsom fluorescens in situ hybridisering (FISH) kan kun analysere DNA ved en opløsning på flere hundrede tusinde basepar.
Reeds nye højhastigheds AFM-metode kan kortlægge DNA til en opløsning på snesevis af basepar, mens der skabes billeder på op til en million basepar i størrelse. Og det gør det ved hjælp af en brøkdel af mængden af prøve, der kræves til DNA -sekventering.
"DNA-sekventering er et kraftfuldt værktøj, men det er stadig ret dyrt og har flere teknologiske og funktionelle begrænsninger, der gør det vanskeligt at kortlægge store områder af genomet effektivt og præcist, " siger Jason Reed, Ph.D., hovedforsker på undersøgelsen. Reed er medlem af forskningsprogrammet Cancer Molecular Genetics ved VCU Massey Cancer Center og lektor i Institut for Fysik ved VCU College of Humanities and Sciences. "Vores tilgang bygger bro mellem DNA-sekventering og andre fysiske kortlægningsteknikker, der mangler opløsning. Den kan bruges som en selvstændig metode, eller den kan supplere DNA-sekventering ved at reducere kompleksitet og fejl, når de små stykker af genomet analyseres under sekventeringsproces."
IBM-forskere skabte overskrifter i 1989, da de udviklede AFM-teknologi og brugte en beslægtet teknik til at omarrangere molekyler på atomniveau for at stave "IBM". AFM opnår dette detaljeringsniveau ved at bruge en mikroskopisk stylus - svarende til en nål på en pladespiller - der næsten ikke kommer i kontakt med overfladen af det materiale, der studeres. Samspillet mellem pennen og molekylerne skaber billedet. Imidlertid, traditionel AFM er for langsom til medicinske anvendelser, og derfor bruges den primært af ingeniører inden for materialevidenskab.
"Vores enhed fungerer på samme måde som AFM, men vi flytter prøven forbi pennen med en meget større hastighed og bruger optiske instrumenter til at opdage interaktionen mellem pennen og molekylerne. Vi kan opnå det samme detaljeringsniveau som traditionel AFM, men kan behandle materiale mere end tusind gange hurtigere, " siger Reed, hvis team beviste, at teknologien kan mainstreames ved at bruge optisk udstyr, der findes i dvd-afspillere. "Højhastigheds AFM er ideel til nogle medicinske applikationer, da det kan behandle materialer hurtigt og give hundredvis af gange mere opløsning end sammenlignelige billeddannelsesmetoder."
At øge hastigheden på AFM var kun en forhindring, som Reed og hans kolleger skulle overvinde. For faktisk at identificere genetiske mutationer i DNA, de skulle udvikle en måde at placere markører eller mærker på overfladen af DNA-molekylerne, så de kunne genkende mønstre og uregelmæssigheder. En genial kemisk stregkodningsløsning blev udviklet ved hjælp af en form for CRISPR-teknologi.
CRISPR har for nylig skabt mange overskrifter med hensyn til genredigering. CRISPR er et enzym, som forskere har været i stand til at "programmere" ved hjælp af målrettet RNA for at skære DNA på præcise steder, som cellen derefter reparerer på egen hånd. Reeds team ændrede de kemiske reaktionsbetingelser for CRISPR-enzymet, så det kun klæber til DNA'et og faktisk ikke skærer det.
"Fordi CRISPR -enzymet er et protein, der er fysisk større end DNA -molekylet, det er perfekt til denne stregkodningsapplikation, "siger Reed." Vi var forbløffet over at opdage, at denne metode er næsten 90 procent effektiv til binding til DNA -molekylerne. Og fordi det er nemt at se CRISPR-proteinerne, du kan spotte genetiske mutationer blandt mønstrene i DNA."
For at demonstrere teknikkens effektivitet, forskerne kortlagde genetiske translokationer i lymfeknudebiopsier af lymfompatienter. Translokationer opstår, når en sektion af DNA'et bliver kopieret og indsat det forkerte sted i genomet. De er især udbredt i blodkræft såsom lymfom, men forekommer også i andre kræftformer.
Selvom der er mange potentielle anvendelser for denne teknologi, Reed og hans team fokuserer på medicinske applikationer. De er i øjeblikket ved at udvikle software baseret på eksisterende algoritmer, der kan analysere mønstre i sektioner af DNA på op til og over en million basepar i størrelse. Når det er afsluttet, det ville ikke være svært at forestille sig dette instrument i skoæskestørrelse i patologiske laboratorier, der hjælper med diagnosticering og behandling af sygdomme forbundet med genetiske mutationer.
 Varme artikler
Varme artikler
-
 En biobank af reversible mutante embryonale stamcellerHaplobank indeholder over 100, 000 muterede, betingede mus embryonale stamcellelinjer, målrettet mod omkring 70% af det proteinkodende genom. Kredit:IzabellaKaminski Videnskabsmand ved IMBA udvikl
En biobank af reversible mutante embryonale stamcellerHaplobank indeholder over 100, 000 muterede, betingede mus embryonale stamcellelinjer, målrettet mod omkring 70% af det proteinkodende genom. Kredit:IzabellaKaminski Videnskabsmand ved IMBA udvikl -
 Forskere viser molekylært grundlag for myrer, der fungerer som livvagter for planterMyreplanten Cordia nodosa har hul, hævede stængler det hus Allomerus octoarticulatus myre livvagter, der beskytter planten mod planteædere. Kredit:G. A. Miller Selvom du måske ikke tænker på m
Forskere viser molekylært grundlag for myrer, der fungerer som livvagter for planterMyreplanten Cordia nodosa har hul, hævede stængler det hus Allomerus octoarticulatus myre livvagter, der beskytter planten mod planteædere. Kredit:G. A. Miller Selvom du måske ikke tænker på m -
 Dino-dræbende asteroide fremskyndede fugleudviklingenEn gennemsnitlig fugl fra umiddelbart før K-Pg-grænsen vejede omkring 1 kg, ligner en Gulnæbbet And (til venstre); en gennemsnitlig fugl vejer i dag omkring 37 g, størrelsen af Cape Weaver (til højr
Dino-dræbende asteroide fremskyndede fugleudviklingenEn gennemsnitlig fugl fra umiddelbart før K-Pg-grænsen vejede omkring 1 kg, ligner en Gulnæbbet And (til venstre); en gennemsnitlig fugl vejer i dag omkring 37 g, størrelsen af Cape Weaver (til højr -
 Hvordan sporer du en hemmelighedsfuld høg? Følg isotoperneHensts goshawk. Kredit:Sarah Karpanty/Virginia Tech Professor ved University of Cincinnati Brooke Crowley ønskede at kende jagtområdet for Hensts goshawk, en stor skovlevende rovfugl, der ligger i
Hvordan sporer du en hemmelighedsfuld høg? Følg isotoperneHensts goshawk. Kredit:Sarah Karpanty/Virginia Tech Professor ved University of Cincinnati Brooke Crowley ønskede at kende jagtområdet for Hensts goshawk, en stor skovlevende rovfugl, der ligger i
- Sådan finder du en vinkel i trigonometri
- Juno-missionen til Jupiter leverer første videnskabelige resultater
- Opbevaring af CO2 under jorden kan bremse kulstofemissioner, men er det sikkert?
- Er det muligt at teste et atomvåben uden at producere radioa
- Interview med en robot:AI -revolution rammer menneskelige ressourcer
- Bæredygtige optiske fibre udviklet af methylcellulose


