Når forskere ikke har de proteiner, de har brug for, kan de få AI til at hallucinere nye strukturer
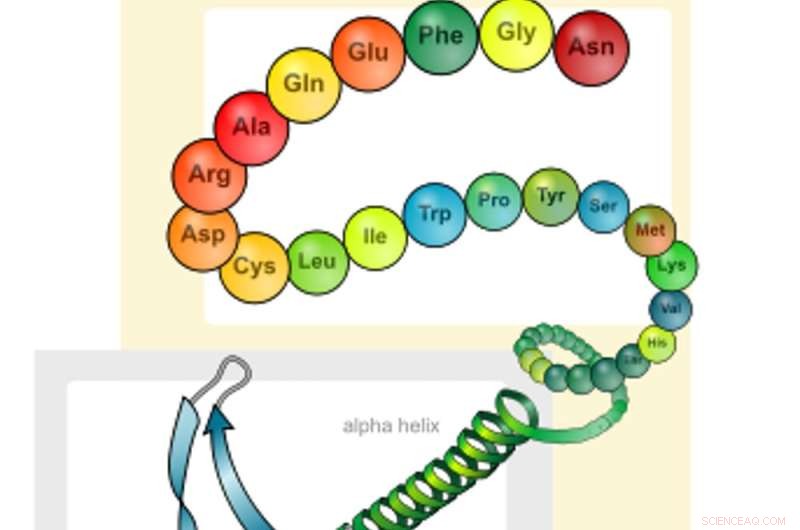
Aminosyrekæder foldes til proteiner. Kredit:LadyofHats/Wikimedia Commons
Alle levende organismer bruger proteiner, som omfatter et stort antal komplekse molekyler. De udfører en bred vifte af funktioner, lige fra at tillade planter at bruge solenergi til iltproduktion til at hjælpe dit immunsystem med at bekæmpe patogener til at lade dine muskler udføre fysisk arbejde. Mange lægemidler er også baseret på proteiner.
For mange områder inden for biomedicinsk forskning og lægemiddeludvikling er der dog ingen naturlige proteiner, der kan tjene som egnede udgangspunkter for at bygge nye proteiner. Forskere, der designer nye lægemidler for at forhindre COVID-19-infektion eller udvikler proteiner, der kan tænde eller slukke for gener eller forvandle celler til computere, var nødt til at skabe nye proteiner fra bunden.
Denne proces med de novo proteindesign kan være svær at få rigtigt. Proteiningeniører som mig har forsøgt at finde ud af måder til mere effektivt og præcist at designe nye proteiner med de egenskaber, vi har brug for.
Heldigvis kan en form for kunstig intelligens kaldet deep learning give en elegant måde at skabe proteiner, der ikke eksisterede tidligere - hallucination.
Design af proteiner fra bunden
Proteiner består af hundredvis til tusindvis af mindre byggesten kaldet aminosyrer. Disse aminosyrer er forbundet med hinanden i lange kæder, der foldes sammen for at danne et protein. Den rækkefølge, hvori disse aminosyrer er forbundet med hinanden, bestemmer hvert proteins unikke struktur og funktion.
Den største udfordring, proteiningeniører står over for, når de designer nye proteiner, er at finde en proteinstruktur, der vil udføre en ønsket funktion. For at omgå dette problem laver forskere typisk designskabeloner baseret på naturligt forekommende proteiner med en lignende funktion. Disse skabeloner har instruktioner om, hvordan man skaber de unikke folder af hvert bestemt protein. Men fordi der skal oprettes en skabelon for hver enkelt fold, er denne strategi tidskrævende, arbejdskrævende og begrænset af, hvilke proteiner der er tilgængelige i naturen.
I løbet af de sidste par år har forskellige forskergrupper, inklusive det laboratorium, jeg arbejder i, udviklet en række dedikerede dybe neurale netværk - computerprogrammer, der bruger flere behandlingslag til at "lære" af inputdata for at komme med forudsigelser om et ønsket output.
Når det ønskede output er et nyt protein, sættes millioner af parametre, der beskriver forskellige facetter af et protein, ind i netværket. Det, der er forudsagt, er en tilfældigt valgt sekvens af aminosyrer, der er kortlagt på den mest sandsynlige 3D-struktur, som sekvensen ville tage.
Netværksforudsigelser for en tilfældig aminosyresekvens er slørede, hvilket betyder, at den endelige struktur af proteinet ikke er særlig entydig, mens både naturligt forekommende proteiner og proteiner bygget fra bunden producerer meget mere veldefinerede proteinstrukturer.
Hallucinerende nye proteiner
Disse observationer antyder én måde, hvorpå nye proteiner kan genereres fra bunden – ved at justere tilfældige input til netværket, indtil forudsigelser giver en veldefineret struktur.
Proteingenereringsmetoden, som mine kolleger og jeg udviklede, ligner konceptuelt computervisionsmetoder som Googles DeepDream, der finder og forbedrer mønstre i billeder.
Disse metoder fungerer ved at tage netværk, der er trænet til at genkende menneskelige ansigter eller andre mønstre i billeder, såsom formen af et dyr eller en genstand, og vende dem om, så de lærer at genkende disse mønstre, hvor de ikke eksisterer. I DeepDream, for eksempel, får netværket vilkårlige inputbilleder, der justeres, indtil netværket kan genkende et ansigt eller en anden form i billedet. Selvom det endelige billede ikke ligner et ansigt for en person, der ser på det, ville det gøre det for det neurale netværk.
Produkterne fra denne teknik omtales ofte som hallucinationer, og det er også det, vi kalder vores designede proteiner.
Vores metode starter med at sende en tilfældig aminosyresekvens gennem et dybt neuralt netværk. De resulterende forudsigelser er oprindeligt slørede med uklare strukturer, som forventet for tilfældige sekvenser. Dernæst introducerer vi en mutation, der ændrer en aminosyre i kæden til en anden og sender denne nye sekvens gennem netværket igen. Hvis denne ændring giver proteinet en mere defineret struktur, så beholder vi aminosyren og introducerer en anden mutation i sekvensen.
For hver gentagelse af denne proces kommer proteinerne tættere og tættere på den rigtige form, de ville tage, hvis de blev produceret i naturen. Der kræves tusindvis af gentagelser for at skabe et helt nyt protein.
Ved hjælp af denne proces genererede vi 2.000 nye proteinsekvenser, der blev forudsagt at folde til veldefinerede strukturer. Af disse udvalgte vi over 100, der var de mest distinkte i form til fysisk at genskabe i laboratoriet. Til sidst valgte vi tre af topkandidaterne til detaljeret analyse og bekræftede, at de var tætte matcher med de former, som vores hallucinerede modeller forudsagde.
Hvorfor hallucinere nye proteiner?
Vores hallucinationstilgang forenkler i høj grad proteindesignpipelinen. Ved at eliminere behovet for skabeloner kan forskere direkte fokusere på at skabe et protein baseret på ønskede funktioner og lade netværket tage sig af at finde ud af strukturen for dem.
Vores arbejde åbner op for flere muligheder for forskere at udforske. Vores laboratorium undersøger i øjeblikket, hvordan man bedst bruger denne hallucinationstilgang til at generere endnu mere specificitet i funktionen af designet proteiner. Vores tilgang kan også let udvides til at designe nye proteiner ved hjælp af andre nyligt udviklede dybe neurale netværk.
De potentielle anvendelser af de novo-proteiner er enorme. Med dybe neurale netværk vil forskere være i stand til at skabe endnu flere proteiner, der kan nedbryde plastik for at reducere miljøforurening, identificere og reagere på usunde celler og forbedre vacciner mod eksisterende og nye patogener – for blot at nævne nogle få.
 Varme artikler
Varme artikler
-
 Crunch tid for fødevaresikkerhedØrkenhoppe. Kredit:© Rothamsted Research Følelser løber ofte højt, når det drejer sig om insekter, med mange mennesker endda klamre til at se på dem, endsige røre eller sluge dem. Og alligevel giv
Crunch tid for fødevaresikkerhedØrkenhoppe. Kredit:© Rothamsted Research Følelser løber ofte højt, når det drejer sig om insekter, med mange mennesker endda klamre til at se på dem, endsige røre eller sluge dem. Og alligevel giv -
 Hvad er de primære funktioner ved fosfolipider?Phospholipider er udbredt i cellerne i bakterier og eukaryoter. De er molekyler lavet af et fosfathoved og en lipidhale. Hovedet betragtes som vandelskende eller hydrofil, mens halen er hydrofob eller
Hvad er de primære funktioner ved fosfolipider?Phospholipider er udbredt i cellerne i bakterier og eukaryoter. De er molekyler lavet af et fosfathoved og en lipidhale. Hovedet betragtes som vandelskende eller hydrofil, mens halen er hydrofob eller -
 Mutation i nøglemolekyler kunne stoppe gonoréinfektionVariante tdfJ-gener udtrykkes fra et inducerbart ektopisk sted i gonokokkromosomet. Vildtype og muterede former af tdfJ blev klonet ind i en komplementeringsvektor designet til at indsætte i et ektopi
Mutation i nøglemolekyler kunne stoppe gonoréinfektionVariante tdfJ-gener udtrykkes fra et inducerbart ektopisk sted i gonokokkromosomet. Vildtype og muterede former af tdfJ blev klonet ind i en komplementeringsvektor designet til at indsætte i et ektopi -
 Daglige pastoratiske aktiviteter påvirker ikke plettede hyæner negativt i TanzaniaPlettet hyæne med Maasai-pastoralist og kvæg i Ngorongoro-krateret. Kredit:Oliver Höner/Leibniz-IZW Pastoralister, der hyrder deres husdyr gennem plettede hyæneklaners territorier ad dedikerede sti
Daglige pastoratiske aktiviteter påvirker ikke plettede hyæner negativt i TanzaniaPlettet hyæne med Maasai-pastoralist og kvæg i Ngorongoro-krateret. Kredit:Oliver Höner/Leibniz-IZW Pastoralister, der hyrder deres husdyr gennem plettede hyæneklaners territorier ad dedikerede sti
- Forsinket langsom havreaktion på kuldioxidfjernelse forårsager asymmetrisk ændring i tropisk nedb…
- Giv slip! Disney streamer til virus-ramte Europa
- Røntgen-Rabi-svingninger mellem kerner observeret i koblede hulrum
- Sådan beregnes euklidisk afstand
- Ny lavprismetode til fremstilling af lysbaserede lab-on-a-chip-enheder til hurtige medicinske tests
- Større er nogle gange bedre, når det kommer til bedriftens størrelse


