Fersken perfektion:Avancerede gennetværk afslører frugtegenskaber
En undersøgelse har udnyttet ferskengenomet og skabt storstilede gen-co-ekspressionsnetværk (GCN'er), der forudsiger genfunktioner og strømliner ferskenavlsprocessen. Denne innovative tilgang løser den komplekse opgave at identificere gener forbundet med ønskelige avlsegenskaber hos ferskner.
Ferskengenetik står over for udfordringer såsom lange intergenerationstider og begrænsede genetiske transformationssystemer, hvilket gør genidentifikation og validering kompleks. Traditionelle tilgange kommer ofte til kort i at forudsige genfunktioner på grund af disse begrænsninger.
Gen-co-ekspressionsnetværk (GCN'er), som kortlægger forholdet mellem genudtryk, tilbyder en lovende løsning. Ved at udnytte "guilty-by-association"-princippet kan GCN'er forudsige genfunktioner baseret på fælles ekspressionsmønstre. På grund af disse udfordringer er der et presserende behov for innovative værktøjer som GCN'er for at forbedre vores forståelse af ferskengenetik.
Forskere fra Institut de Recerca i Tecnologia Agroalimentàries (IRTA) og Centre de Recerca en Agrigenòmica (CRAG) offentliggjorde en undersøgelse i tidsskriftet Horticulture Research den 2. januar 2024. Undersøgelsen introducerer et nyt værktøj, der udnytter GCN'er til at forudsige genfunktioner i ferskner, hvilket potentielt revolutionerer frugtavl.
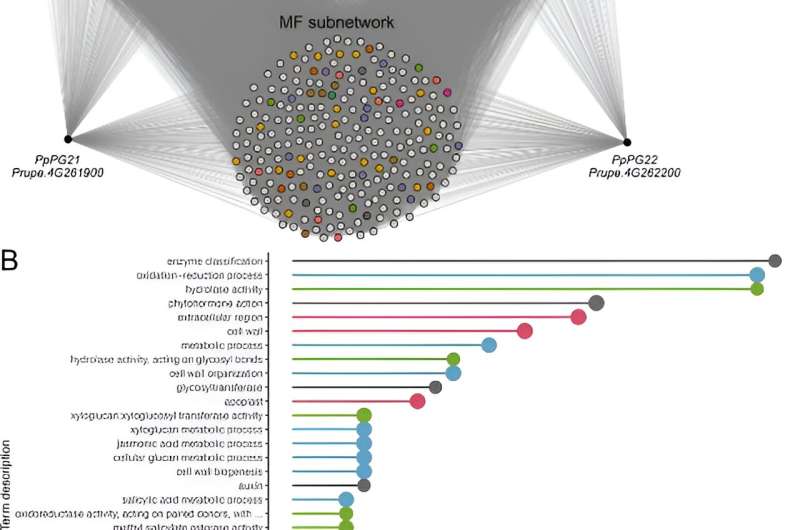
Undersøgelsen skabte fire GCN'er fra 604 Illumina RNA-Seq-biblioteker og evaluerede deres ydeevne ved hjælp af en maskinlæringsalgoritme baseret på "guilty-by-association"-princippet. Blandt netværkene viste COO300 den bedste ydeevne og omfattede 21.956 gener. For at validere COO300 gennemførte forskere to casestudier.
I det første casestudie blev generne PpPG21 og PpPG22, involveret i blødgøring af ferskenfrugter, analyseret. De co-udtrykte gener dannede det smeltende kød (MF) netværk, som blev beriget med udtryk relateret til cellevægsorganisation og fytohormonsignalering. MF-netværket inkluderede gener forbundet med cellevægsadskillelse og modningsrelaterede fytohormoner, hvilket demonstrerer COO300's evne til at forudsige genfunktioner nøjagtigt.
I det andet casestudie blev transkriptionsfaktoren PpMYB10.1, som regulerer anthocyaninakkumulering, undersøgt. Frugtfarve-netværket (FC) identificerede gener involveret i anthocyaninmetabolisme og -regulering. Ved at sammenligne FC-netværket med vinortologer identificerede forskere bevarede regulatoriske netværk, hvilket yderligere validerede COO300's forudsigelsesevne.
Dr. Iban Eduardo, en førende forsker ved CRAG, udtalte:"Udviklingen af COO300-netværket repræsenterer et betydeligt gennembrud inden for ferskengenetik. Ved nøjagtigt at forudsige genfunktioner forbedrer dette værktøj ikke kun vores forståelse af ferskenbiologi, men baner også vejen for mere målrettede og effektive avlsstrategier."
Undersøgelsen tilbyder opdrættere et kraftfuldt værktøj til at forbedre ferskensorter. Ved at afkode genfunktioner forventes PeachGCN v1.0 og dets scripts, tilgængelige for alle, at drive fremskridt inden for smag, levetid og ernæring af ferskner. Denne genomiske indsigt er en varsel om forandring, der signalerer et skift i retning af datadrevet avl i landbruget.
Flere oplysninger: Felipe Pérez de los Cobos et al., Udforskning af gen-samekspressionsnetværk i stor skala i fersken (Prunus persica L.):et nyt værktøj til at forudsige genfunktion, Harticulture Research (2024). DOI:10.1093/time/uhad294
Journaloplysninger: Gavebrugsforskning
Leveret af NanJing Agricultural University
 Varme artikler
Varme artikler
-
 Undervandsvarmeinferno hærger middelhavskorallerDenne sommer ramte en stor marin hedebølge det vestlige Middelhav med vand op til fem grader celsius varmere end normalt. I Middelhavets tempererede lavvandede områder står engang levende røde og l
Undervandsvarmeinferno hærger middelhavskorallerDenne sommer ramte en stor marin hedebølge det vestlige Middelhav med vand op til fem grader celsius varmere end normalt. I Middelhavets tempererede lavvandede områder står engang levende røde og l -
 Hvordan svømmer vandskimmelsporer?Til venstre:Billede i falsk farve af zoosporen af en oomycete (Phytophthora parasitica). Bemærk, at den forreste flagel har udseende af tinsel, mens den bagerste flagel er glat. Til højre:Højhastigh
Hvordan svømmer vandskimmelsporer?Til venstre:Billede i falsk farve af zoosporen af en oomycete (Phytophthora parasitica). Bemærk, at den forreste flagel har udseende af tinsel, mens den bagerste flagel er glat. Til højre:Højhastigh -
 Kyllinger med rød glente, der er født under tørkeperioder, er arrede for livet:Den skjulte trusse…Kredit:Werner Baumgarten/Shutterstock Tag en køretur ned ad Englands M40-motorvej, og chancerne er, at du vil se en stor fugl med en kløvet hale over hovedet, der leder efter roadkill:den røde glen
Kyllinger med rød glente, der er født under tørkeperioder, er arrede for livet:Den skjulte trusse…Kredit:Werner Baumgarten/Shutterstock Tag en køretur ned ad Englands M40-motorvej, og chancerne er, at du vil se en stor fugl med en kløvet hale over hovedet, der leder efter roadkill:den røde glen -
 Coyotes er kommet for at blive i nordamerikanske byer. Sådan værdsætter du dem på afstandKredit:Unsplash/CC0 Public Domain Coyoter er blevet praktisk talt allestedsnærværende i de nedre 48 USA, og de dukker i stigende grad op i byer. Tiltrækningerne er rigelig mad og grønne områder i b
Coyotes er kommet for at blive i nordamerikanske byer. Sådan værdsætter du dem på afstandKredit:Unsplash/CC0 Public Domain Coyoter er blevet praktisk talt allestedsnærværende i de nedre 48 USA, og de dukker i stigende grad op i byer. Tiltrækningerne er rigelig mad og grønne områder i b
- Metal-organiske forbindelser producerer en ny klasse af glas
- Hvad er ulemperne ved HPLC?
- Ny tilgang kunne hurtigt identificere de bedste organiske solcelleblandinger
- Hacking evolution, screeningsteknik kan forbedre det mest udbredte enzym
- Droner tæller omkostningerne ved 2017 f.Kr. naturbrande
- Fremmer fremtiden:Det mindste helt digitale kredsløb åbner døre til 5 nm næste generations halvl…


