Behov for hastighed gør genomredigering effektiv, hvis ikke bedre
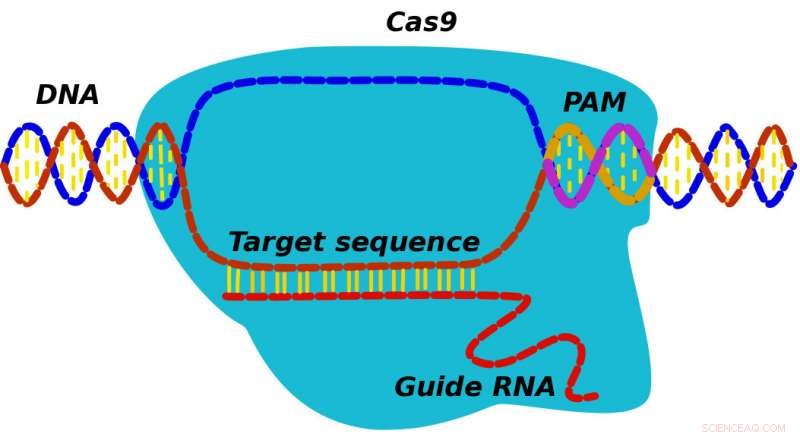
Når et CRISPR-Cas9-protein finder sit mål, den finder først en spacersekvens kendt som PAM og søger derefter tilstødende DNA for at se, om den matcher Cas9's guide RNA. En ny model udviklet af forskere ved Rice University kunne hjælpe med at afdække detaljer om den mekanisme, hvormed CRISPR-Cas9 kan erstatte mutationer med korrekt DNA. Kredit:Alexey Shvets/Rice University
Rice University forskere har udviklet en beregningsmodel til at kvantificere den mekanisme, hvormed CRISPR-Cas9 proteiner finder deres genomredigeringsmål.
Anatoly Kolomeisky, en risprofessor i kemi og kemisk og biomolekylær teknik, og alumnen Alexey Shvets tilpassede et system, de udviklede tidligere for at vise, hvordan proteiner generelt finder deres biologiske mål. De håber, at den reviderede model vil hjælpe med at låse op for de resterende mysterier ved CRISPR.
I sin naturlige tilstand, CRISPR, som står for "klyngede regelmæssigt mellemliggende korte palindromiske gentagelser, "er den biologiske mekanisme, hvormed bakterier beskytter sig mod virusinfektioner. Bakterierne inkorporerer en kopi af det fremmede DNA og bygger en registrering af alle dem, der invaderer. De refererer til den rekord, når nye invaders opdages og bruger den til at ødelægge dem.
I de seneste år, forskere er begyndt at tilpasse mekanismen til brug ved genomredigering, som har potentiale til at helbrede sygdom og forbedre organismer, herunder mennesker. Men en anstødssten har været risikoen for, at CRISPR-Cas9-proteiner, et af de systemer, der anvender CRISPR -tilgangen, vil klippe og erstatte de forkerte målsekvenser, introducerer mutationer.
Rismodellen beskrevet i Biofysisk Journal fandt det sandsynligt, at CRISPR-Cas9 lokaliserer gode mål mere effektivt, når disse redigeringer uden for mål får lov til at ske, fordi proteinerne ikke spilder tid med at adskille sig fra off-goals til at fortsætte søgningen.
Det er måske en god ting, eller måske ikke men det er bestemt værd at studere, Sagde Kolomeisky.
"Fejlprocenten (off-target-skæring) er undertiden 10-20 procent, "sagde han." Vi har to ideer om dette:Den ene er, at vira muterer meget hurtigt, og måske prøver bakterier at skære mål, der kun er lidt muterede som en måde at være mere fleksibel på. Den anden er, at der er proteiner, der kan rette fejl, så hvis der ikke er mange forkerte nedskæringer, systemet kan tåle dem.
Kolomeisky sagde, at hans model er et simpelt skridt i retning af at finde ud af dynamikken i CRISPR -redigering. "CRISPR-Cas9 er den mest populære variant, fordi den kun har et protein og er lettere, biologisk, at arbejde med, " han sagde.
Rice -laboratoriet udviklede sin originale model for at lære, hvordan proteiner glider langs DNA for at finde mål og udløse processer som gentranskription. Kolomeisky bemærkede, at CRISPR-pioneren Jennifer Doudna opdagede, at CRISPR-Cas9 ikke søger på samme måde. "Hun fandt ud af, at det ikke glider nogen steder på DNA'et, " han sagde.
I stedet, ifølge Doudna og hendes team, proteinet genkender oprindeligt tre-nukleotid PAM (for protospacer tilstødende motiv) sekvenser, der markerer placeringen af potentielle mål. "CRISPR finder og binder til PAM, og derefter undersøger dets associerede RNA det tilstødende DNA for at se, om dette er målet, "Sagde Kolomeisky." Hvis det er, proteinet begynder at skære. Hvis ikke, det adskiller sig og leder andre steder. "
I Doudnas efterfølgende eksperimenter med PAM -sekvenser fjernet, CRISPR-Cas9 proteiner kunne slet ikke finde deres mål. Så PAM'er har en vigtig rolle og er ikke bare en generisk afstandsstykke, han sagde. "Så snart jeg læste dette, Jeg forstod, at vi også kunne bruge vores model her. "
Den teoretiske model ser på processer ved første passage-dem der sker, når et system krydser en fysisk eller kemisk tærskel, som at finde en relevant PAM-for at spore CRISPR-Cas9-proteiner indsat i en celle, da de først undersøger PAM-sekvenser og derefter, mens bundet til PAM'er, søg efter DNA -målet, der matcher Cas9's RNA.
De fandt CRISPR'er, der undgår nedskæringer uden for mål ved at adskille sig fra "forkert" DNA, tager længere tid at afregne end en, der blot skærer off-mål. "At gå til den forkerte PAM tager tid, "Kolomeisky sagde." Vores beregning viser, at CRISPR hurtigere kan finde rigtige mål, når det nogle gange skærer de forkerte steder. Den brøkdel, der går til de rigtige mål, kan være mindre, men du vil til sidst klippe dem.
"Det er en simpel model og præcist opløselig, "Sagde Kolomeisky." Hvis nogen vil teste, modellen kan give specifikke forudsigelser og i nogle tilfælde tilbyde tendenser for det, der skal observeres. "Det, der mangler i modellen, er evnen til at se, om RNA -nøglen genkender sit mål samtidigt - bindende til DNA på én gang - eller sekventielt, nukleotid ved nukleotid.
"Det mest imponerende ved CRISPR er ikke opdagelsen af et immunsystem i bakterier, men det faktum, at dette har skabt en revolution inden for bioteknologi, fordi det betyder, at vi i enhver celle kan skære ethvert DNA på et bestemt sted, meget præcist, "Sagde Kolomeisky." Jeg håber, at vores arbejde vil stimulere mere grundlæggende undersøgelser, fordi jeg godt kan lide CRISPR -metoden. Men jeg er ikke glad, når folk anvender det uden at forstå, hvordan det fungerer på molekylært niveau. "
Shvets er nu postdoktor ved Massachusetts Institute of Technology. Kolomeisky er professor i kemi og kemisk og biomolekylær teknik.
 Varme artikler
Varme artikler
-
 Hvorfor vi ikke skal være for hurtige til at give migrerende dyr skylden for global sygdomDen rødhalsede stint er meget udsat for sedimentmikrober, når den fouragerer efter de mikroskopiske hvirvelløse dyr, der giver næring til dens enorme migrationer. Forfatter angivet Er du nogensind
Hvorfor vi ikke skal være for hurtige til at give migrerende dyr skylden for global sygdomDen rødhalsede stint er meget udsat for sedimentmikrober, når den fouragerer efter de mikroskopiske hvirvelløse dyr, der giver næring til dens enorme migrationer. Forfatter angivet Er du nogensind -
 Malariaparasit pakker genetisk materiale til rejse fra myg til menneskerFordi malariaparasitten Plasmodium ikke kan forudse, hvornår den kan overføres fra en myg til en pattedyrvært, det bruger specialiserede poly(A)-bindende proteiner til at pakke og beskytte dets geneti
Malariaparasit pakker genetisk materiale til rejse fra myg til menneskerFordi malariaparasitten Plasmodium ikke kan forudse, hvornår den kan overføres fra en myg til en pattedyrvært, det bruger specialiserede poly(A)-bindende proteiner til at pakke og beskytte dets geneti -
 Videnskabelig evaluering af næsehornsdiæter forbedrer zooHvide næsehorn tæt på Waterberg National Park, Namibia. Kredit:Wikipedia/GFDL 1.2 En nyligt offentliggjort undersøgelse i tidsskriftet Pachyderm fremhæver den igangværende indsats fra akkrediter
Videnskabelig evaluering af næsehornsdiæter forbedrer zooHvide næsehorn tæt på Waterberg National Park, Namibia. Kredit:Wikipedia/GFDL 1.2 En nyligt offentliggjort undersøgelse i tidsskriftet Pachyderm fremhæver den igangværende indsats fra akkrediter -
 Nytænkning af transkriptionsfaktorer og genekspressionDNA-løkker skaber, hvad Whitehead Institute-forskere kalder isolerede kvarterer for gener og deres promotorer og forstærkere. Kredit:Steven Lee/Whitehead Institute Transkription - læsning af et se
Nytænkning af transkriptionsfaktorer og genekspressionDNA-løkker skaber, hvad Whitehead Institute-forskere kalder isolerede kvarterer for gener og deres promotorer og forstærkere. Kredit:Steven Lee/Whitehead Institute Transkription - læsning af et se
- Sådan adskilles alkohol fra vand
- En ny tilgang til fremstilling af løsningsbearbejdelige 2D-halvledere
- Politisk videnskabsmand beskriver stigningen i negativ partiskhed, og hvordan det driver vælgerne
- Instagram kræver fødselsdatoer for at blokere mindreåriges brug
- Billeder af bisonknogler giver et vindue til de kulturelle og økologiske relationer, der binder dyr…
- Syv moralske regler findes over hele verden


