Undersøgelse af planteenzym viser, at proteiner kan ændre deres strukturelle arrangement med overraskende lethed
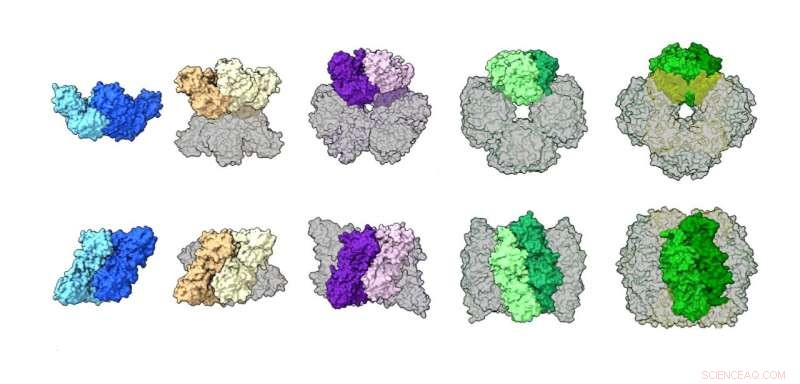
En sammenligning af rubisco-samlinger fra forskellige arter, der illustrerer en bred vifte af strukturer, herunder en dimer, tetramer, hexamer, octamer og hexadecamer (16 enheder protein). Kredit:Shih Lab/Berkeley Lab
Når du tænker på proteiner – enzymerne, signalmolekylerne og de strukturelle komponenter i alt levende – tænker du måske på enkeltstrenge af aminosyrer, organiseret som perler på en snor. Men næsten alle proteiner består af flere strenge, der er foldet op og bundet til hinanden, og danner komplicerede 3D-superstrukturer kaldet molekylære samlinger. Et af de vigtigste trin til at forstå biologi er at opdage, hvordan et protein gør sit arbejde, hvilket kræver viden om dets strukturer ned til atomniveau.
I løbet af det seneste århundrede har videnskabsmænd udviklet og implementeret fantastiske teknologier såsom røntgenkrystallografi og kryo-elektronmikroskopi til at bestemme proteinstrukturen og derved besvaret utallige vigtige spørgsmål. Men nyt arbejde viser, at forståelsen af proteinstruktur nogle gange kan være mere kompliceret, end vi tror.
En gruppe forskere fra Lawrence Berkeley National Laboratory (Berkeley Lab), der studerede verdens mest rigelige protein, et enzym involveret i fotosyntese kaldet rubisco, viste, hvordan evolution kan føre til en overraskende mangfoldighed af molekylære samlinger, der alle udfører den samme opgave. Resultaterne, offentliggjort i dag i Science Advances , afslører muligheden for, at mange af de proteiner, vi troede, vi vidste, faktisk eksisterer i andre, ukendte former.
Historisk set, hvis videnskabsmænd løste en struktur og bestemte, at et protein var dimerisk (sammensat af to enheder), kunne de for eksempel antage, at lignende proteiner også eksisterede i en dimer form. Men lille prøvestørrelse og prøveudtagningsbias – uundgåelige faktorer, da det er meget vanskeligt at omdanne naturligt flydende proteiner til faste, krystalliserede former, der kan undersøges via røntgenkrystallografi – tilslørede virkeligheden.

Albert Lui (til venstre) og Patrick Shih demonstrerer, hvordan de studerede strukturen og udviklingen af rubisco, enzymet, som planter bruger til at udnytte CO2 til at skabe sukker, i Koshland Hall ved UC Berkeley. Kredit:Marilyn Sargent/Berkeley Lab
"Det er ligesom, at hvis du gik udenfor og så nogen gå tur med deres hund, hvis du aldrig havde set en hund før og så en wienerhund, ville du tænke:'OK, sådan ser alle hunde ud." Men det, du skal gøre, er at gå til hundeparken og se al den mangfoldighed af hunde, der er der," sagde hovedforfatter Patrick Shih, en fakultetsforsker i Biosciences Area og direktør for Plant Biosystems Design ved Joint BioEnergy Institute (JBEI). "Et takeaway fra dette papir, der går ud over rubisco, til alle proteiner, er spørgsmålet om, hvorvidt vi ser den sande række af strukturer i naturen, eller om disse skævheder får det til at se ud som om alt ligner en wienerhund."
I håb om at udforske alle de forskellige rubisco-arrangementer i den metaforiske hundepark og lære, hvor de kom fra, samarbejdede Shihs laboratorium med strukturbiologiske eksperter i Bioscience Area ved at bruge Berkeley Labs Advanced Light Source. Sammen studerede holdet en type rubisco (form II) fundet i bakterier og en undergruppe af fotosyntetiske mikrober ved hjælp af traditionel krystallografi - en teknik, der er i stand til opløsning på atomniveau - kombineret med en anden struktur-løsningsteknik, småvinklet røntgenspredning (SAXS), der har lavere opløsning, men kan tage snapshots af proteiner i deres oprindelige form, når de er i flydende blandinger. SAXS har den yderligere fordel ved høj-throughput-kapacitet, hvilket betyder, at den kan behandle snesevis af individuelle proteinsamlinger hurtigt efter hinanden.
Tidligere arbejde havde vist, at den bedre undersøgte type rubisco fundet i planter (form I) altid tager en "oktamerisk kerne"-samling af otte store proteinenheder arrangeret med otte små enheder, hvorimod form II blev antaget at eksistere mest som en dimer med en få sjældne eksempler på hexamerer med seks enheder. Efter at have brugt disse komplementære teknikker til at undersøge prøver af rubisco fra en bred vifte af mikrobearter, observerede forfatterne, at de fleste form II rubisco-proteiner faktisk er hexamerer, med lejlighedsvis dimer, og de opdagede en aldrig før set tetramer (fire enheder) montage.
Ved at kombinere disse strukturelle data med de respektive proteinkodende gensekvenser tillod holdet at udføre forfædres sekvensrekonstruktion - en computerbaseret molekylær evolutionsmetode, der kan estimere, hvordan forfædres proteiner så ud baseret på sekvensen og udseendet af moderne proteiner, der udviklede sig fra dem.
Albert Liu tjekkede nogle dyrkningsplader af E. coli med generne for at lave form II rubisco. Kredit:Albert Liu
Rekonstruktionen antyder, at genet for form II rubisco har ændret sig i løbet af sin evolutionære historie for at producere proteiner med en række strukturer, der transformerer sig til nye former eller vender tilbage til ældre strukturer ret nemt. I modsætning hertil førte selektive tryk i løbet af evolutionen til en række ændringer, der låste form I rubisco på plads - en proces kaldet strukturel forankring - hvilket er grunden til, at den oktameriske samling er det eneste arrangement, vi ser nu. Ifølge forfatterne blev det antaget, at de fleste proteinsamlinger blev forankret over tid af selektivt pres for at forfine deres funktion, som vi ser med form I rubisco. Men denne forskning tyder på, at evolution også kan favorisere fleksible proteiner.
"Det store fund fra dette papir er, at der er meget strukturel plasticitet," sagde Shih, som også er adjunkt ved UC Berkeley. "Proteiner kan være meget mere fleksible på tværs af feltet, end vi har troet."
Efter at have afsluttet den forfædres sekvensrekonstruktion udførte holdet mutationseksperimenter for at se, hvordan ændring af rubisco-samlingen, i dette tilfælde at bryde en hexamer til en dimer, påvirkede enzymets aktivitet. Uventet producerede denne inducerede mutation en form for rubisco, der er bedre til at udnytte sit målmolekyle, CO2 . Al naturligt forekommende rubisco binder ofte O2 af samme størrelse molekyle ved et uheld, hvilket sænker enzymets produktivitet. Der er stor interesse i at genmodificere rubiscoen i landbrugsplantearter for at øge proteinets affinitet for CO2 , for at producere mere produktive og ressourceeffektive afgrøder. Der har dog været meget fokus på proteinets aktive sted – det område af proteinet, hvor CO2 eller O2 bind.
"Dette er en interessant indsigt for os, fordi det antyder, at for at få mere frugtbare resultater, der manipulerer rubisco, kan vi ikke bare se på det enkleste svar, den region af enzymet, der faktisk interagerer med CO2 ," sagde førsteforfatter Albert Liu, en kandidatstuderende i Shihs laboratorium. "Måske er der mutationer uden for det aktive sted, som faktisk deltager i denne aktivitet og potentielt kan ændre proteinfunktionen på en måde, som vi ønsker. Så det er noget, der virkelig åbner døre til fremtidige forskningsmuligheder."
Medforfatter Paul Adams, Associate Laboratory Director for Biosciences og Vice President for Technology hos JBEI tilføjede:"Blandingen af anvendte teknikker og holdets tværfaglige karakter var en reel nøgle til succes. Arbejdet fremhæver styrken ved at kombinere genomiske data og strukturbiologiske metoder til at studere et af de vigtigste problemer i biologi og nå frem til nogle uventede konklusioner." + Udforsk yderligere
Undersøgelse finder "missing link" i den evolutionære historie af kulstoffikserende protein rubisco
 Varme artikler
Varme artikler
-
 Biologisk ur fundet i svampeparasitter kaster mere lys over zombimyrer -fænomenetInficeret myre, der har låst sig fast på et stykke vegetation efter at være blevet manipuleret af parasitten. Kredit:Brianna Santamaria Charissa de Bekker, Ph.d., kom til UCF tidligere på året for
Biologisk ur fundet i svampeparasitter kaster mere lys over zombimyrer -fænomenetInficeret myre, der har låst sig fast på et stykke vegetation efter at være blevet manipuleret af parasitten. Kredit:Brianna Santamaria Charissa de Bekker, Ph.d., kom til UCF tidligere på året for -
 Forskere identificerer gen, der deltager i bladreaktion på miljøforholdGenetiske beviser bekræftede, at HdSTM deltager i reguleringen af heterofyli. Kredit:IHB Heterophylly, bladformens plasticitet som reaktion på miljøforhold, forekommer i vand- og amfibiske plante
Forskere identificerer gen, der deltager i bladreaktion på miljøforholdGenetiske beviser bekræftede, at HdSTM deltager i reguleringen af heterofyli. Kredit:IHB Heterophylly, bladformens plasticitet som reaktion på miljøforhold, forekommer i vand- og amfibiske plante -
 Telofase: Hvad sker der i denne fase af mitose og meiose?Celledeling er en ekstremt vigtig del i udviklingen af alle celler i alle organismer, inklusive mennesker, dyr og planter. Telofase er det sidste trin i celledeling, før cytokinesis opstår for at
Telofase: Hvad sker der i denne fase af mitose og meiose?Celledeling er en ekstremt vigtig del i udviklingen af alle celler i alle organismer, inklusive mennesker, dyr og planter. Telofase er det sidste trin i celledeling, før cytokinesis opstår for at -
 Hvorfor lyver folk om ting, der let kan modbevises?Hvorfor lyver folk om ting, du nemt kan kontrollere? Ego og ambition. Wavebreakmedia/ThinkStock Det er en ting at fudge om størrelsen på den lækre bas, du fangede, eller hvor meget du vejer på dit kø
Hvorfor lyver folk om ting, der let kan modbevises?Hvorfor lyver folk om ting, du nemt kan kontrollere? Ego og ambition. Wavebreakmedia/ThinkStock Det er en ting at fudge om størrelsen på den lækre bas, du fangede, eller hvor meget du vejer på dit kø
- Sådan laver du din egen køler som en videnskabsprojekt
- Juryen fortæller, at Samsung skal betale stort for at kopiere iPhone-design
- Banebrydende røntgenoptik vil muliggøre fremtidige observatorier
- Maskiner kan ikke personliggøre uddannelse, kun mennesker kan
- Sundhedsplejen har et stort miljømæssigt fodaftryk, hvilket så skader helbredet. Dette er et spø…
- Nedlukning af emissioner vil ikke have nogen effekt på klimaet


