AI-værktøj skaber syntetiske billeder af celler til forbedret mikroskopianalyse
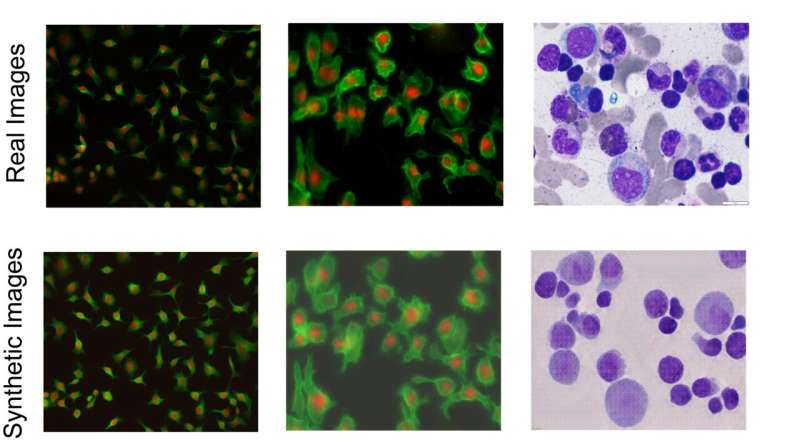
At observere individuelle celler gennem mikroskoper kan afsløre en række vigtige cellebiologiske fænomener, der ofte spiller en rolle i menneskelige sygdomme, men processen med at skelne enkelte celler fra hinanden og deres baggrund er ekstremt tidskrævende - og en opgave, der er velegnet for AI-hjælp.
AI-modeller lærer, hvordan man udfører sådanne opgaver ved at bruge et sæt data, der er kommenteret af mennesker, men processen med at skelne celler fra deres baggrund, kaldet "enkeltcellesegmentering", er både tidskrævende og besværlig. Som et resultat er der en begrænset mængde annoterede data til brug i AI-træningssæt. UC Santa Cruz forskere har udviklet en metode til at løse dette ved at bygge en mikroskopi-billedgenerering AI-model til at skabe realistiske billeder af enkeltceller, som derefter bruges som "syntetiske data" til at træne en AI-model til bedre at udføre enkeltcelle-segmentering.
Den nye software er beskrevet i et nyt papir offentliggjort i tidsskriftet iScience . Projektet blev ledet af adjunkt i biomolekylær teknik Ali Shariati og hans kandidatstuderende Abolfazl Zargari. Modellen, kaldet cGAN-Seg, er frit tilgængelig på GitHub.
"Billederne, der kommer ud af vores model, er klar til at blive brugt til at træne segmenteringsmodeller," sagde Shariati. "På en måde laver vi mikroskopi uden mikroskop, idet vi er i stand til at generere billeder, der er meget tæt på rigtige billeder af celler med hensyn til de morfologiske detaljer af den enkelte celle. Det smukke ved det er, at når de kommer ud af modellen, er de allerede kommenteret og mærket. Billederne viser et væld af ligheder med rigtige billeder, som så giver os mulighed for at generere nye scenarier, som ikke er blevet set af vores model under træningen."
Billeder af individuelle celler set gennem et mikroskop kan hjælpe videnskabsmænd med at lære om celleadfærd og dynamik over tid, forbedre sygdomsdetektion og finde ny medicin. Subcellulære detaljer, såsom tekstur, kan hjælpe forskere med at besvare vigtige spørgsmål, såsom om en celle er kræftfremkaldende eller ej.
Manuelt at finde og mærke cellernes grænser ud fra deres baggrund er dog ekstremt vanskeligt, især i vævsprøver, hvor der er mange celler i et billede. Det kan tage forskere flere dage at udføre manuelt cellesegmentering på kun 100 mikroskopibilleder.
Dyb læring kan fremskynde denne proces, men et indledende datasæt af kommenterede billeder er nødvendigt for at træne modellerne - mindst tusindvis af billeder er nødvendige som basis for at træne en nøjagtig dyb læringsmodel. Selvom forskerne kan finde og kommentere 1.000 billeder, indeholder disse billeder muligvis ikke den variation af funktioner, der vises på tværs af forskellige eksperimentelle forhold.
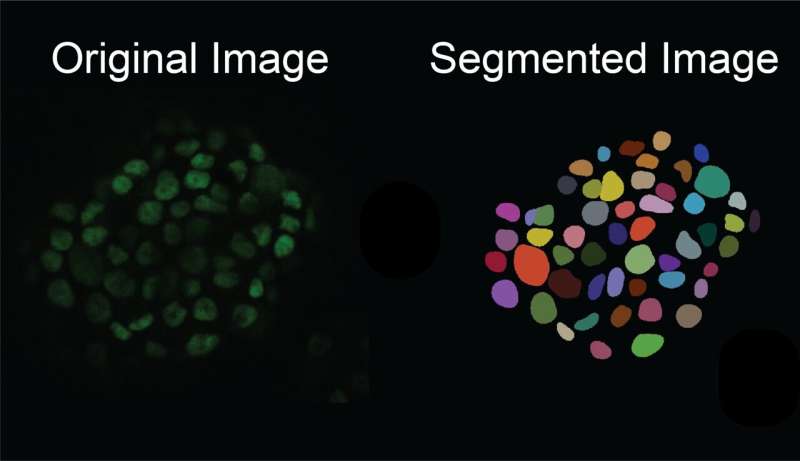
"Du vil gerne vise, at din dybe læringsmodel fungerer på tværs af forskellige prøver med forskellige celletyper og forskellige billedkvaliteter," sagde Zargari. "Hvis du for eksempel træner din model med billeder af høj kvalitet, vil den ikke være i stand til at segmentere cellebillederne af lav kvalitet. Vi kan sjældent finde et så godt datasæt i mikroskopifeltet."
For at løse dette problem skabte forskerne en billed-til-billede-generativ AI-model, der tager et begrænset sæt af kommenterede, mærkede cellebilleder og genererer flere, der introducerer mere indviklede og varierede subcellulære funktioner og strukturer for at skabe et mangfoldigt sæt af "syntetiske" billeder. De kan især generere kommenterede billeder med en høj tæthed af celler, som er særligt vanskelige at annotere i hånden og er særligt relevante til at studere væv. Denne teknik fungerer til at behandle og generere billeder af forskellige celletyper såvel som forskellige billeddannelsesmodaliteter, såsom dem taget ved hjælp af fluorescens eller histologisk farvning.
Zargari, der ledede udviklingen af den generative model, brugte en almindeligt brugt AI-algoritme kaldet et "cyklusgenerativt modstridende netværk" til at skabe realistiske billeder. Den generative model er forbedret med såkaldte "augmentation functions" og et "style injecting network", som hjælper generatoren med at skabe en bred vifte af højkvalitets syntetiske billeder, der viser forskellige muligheder for, hvordan cellerne kunne se ud. Så vidt forskernes viden er det første gang, stilinjiceringsteknikker er blevet brugt i denne sammenhæng.
Derefter bruges dette mangfoldige sæt syntetiske billeder skabt af generatoren til at træne en model til nøjagtigt at udføre cellesegmentering på nye, rigtige billeder taget under eksperimenter.
"Ved at bruge et begrænset datasæt kan vi træne en god generativ model. Ved at bruge den generative model er vi i stand til at generere et mere forskelligartet og større sæt af kommenterede, syntetiske billeder. Ved at bruge de genererede syntetiske billeder kan vi træne en god segmenteringsmodel— det er hovedideen," sagde Zagari.
Forskerne sammenlignede resultaterne af deres model ved hjælp af syntetiske træningsdata med mere traditionelle metoder til træning af AI til at udføre cellesegmentering på tværs af forskellige typer celler. De fandt ud af, at deres model producerer væsentligt forbedret segmentering sammenlignet med modeller trænet med konventionelle, begrænsede træningsdata. Dette bekræfter forskerne, at tilvejebringelse af et mere forskelligartet datasæt under træning af segmenteringsmodellen forbedrer ydeevnen.
Gennem disse forbedrede segmenteringsevner vil forskerne være i stand til bedre at detektere celler og studere variabiliteten mellem individuelle celler, især blandt stamceller. I fremtiden håber forskerne at bruge den teknologi, de har udviklet, til at bevæge sig ud over stillbilleder til at generere videoer, som kan hjælpe dem med at finde ud af, hvilke faktorer der påvirker en celles skæbne tidligt i dens liv og forudsige deres fremtid.
"Vi genererer syntetiske billeder, der også kan omdannes til en time lapse-film, hvor vi kan generere cellers usete fremtid," sagde Shariati. "Med det vil vi se, om vi er i stand til at forudsige en celles fremtidige tilstande, som om cellen skal vokse, migrere, differentiere eller dele sig."
Flere oplysninger: Abolfazl Zargari et al., Forbedret cellesegmentering med begrænsede træningsdatasæt ved hjælp af Cycle Generative Adversarial Networks, iScience (2024). DOI:10.1016/j.isci.2024.109740
Journaloplysninger: iScience
Leveret af University of California - Santa Cruz
Sidste artikelEn lille faktor har stor indflydelse på genomredigering
Næste artikelTryk i livmoderen kan påvirke ansigtets udvikling
 Varme artikler
Varme artikler
-
 Det største dinosaurskelet i Europa er muligvis blevet fundet i PortugalUdgravningskampagnen på det palæontologiske sted Monte Agudo (Pombal, Portugal) resulterede i udvindingen af en del af det fossiliserede skelet af en stor sauropoddinosaur. Kredit:Instituto Dom Luiz
Det største dinosaurskelet i Europa er muligvis blevet fundet i PortugalUdgravningskampagnen på det palæontologiske sted Monte Agudo (Pombal, Portugal) resulterede i udvindingen af en del af det fossiliserede skelet af en stor sauropoddinosaur. Kredit:Instituto Dom Luiz -
 Ny forskning afslører, at kun engelske flaskehalsdelfiner er bosiddende i EnglandKredit:University of Plymouth Den sydvestlige del af England er kendt som et hotspot for hvaler, men indtil for nylig var der lidt kendt om flaskefugle delfiner, der glimtede ud for kysten. Nu af
Ny forskning afslører, at kun engelske flaskehalsdelfiner er bosiddende i EnglandKredit:University of Plymouth Den sydvestlige del af England er kendt som et hotspot for hvaler, men indtil for nylig var der lidt kendt om flaskefugle delfiner, der glimtede ud for kysten. Nu af -
 Strukturel indsigt i processer på DNA-skadestederTil-skala sammensat strukturel model af Mec1-Ddc2-RPA-samlingen og aktivering på DNA-skadesteder. Kredit:Friedrich Miescher Institut for Biomedicinsk Forskning Ishan Deshpande og hans kolleger fra
Strukturel indsigt i processer på DNA-skadestederTil-skala sammensat strukturel model af Mec1-Ddc2-RPA-samlingen og aktivering på DNA-skadesteder. Kredit:Friedrich Miescher Institut for Biomedicinsk Forskning Ishan Deshpande og hans kolleger fra -
 Forskere tager temperaturen af denguefeber risikoKredit:Tommaso Chiodo Når sygdomsbærende myg udvider sig til nye levesteder, offentlige sundhedsembedsmænd bør teste nye ankomsters evne til at overføre vira ved forskellige temperaturer, en ny Ya
Forskere tager temperaturen af denguefeber risikoKredit:Tommaso Chiodo Når sygdomsbærende myg udvider sig til nye levesteder, offentlige sundhedsembedsmænd bør teste nye ankomsters evne til at overføre vira ved forskellige temperaturer, en ny Ya
- 2-D spintronics har allerede transformeret computing-nu fik det til at fungere i tre dimensioner
- Forskere går ind på en potentiel antropocæn gylden spids
- Nye katalysatorer gør effektiv brug af ædle metaller
- Små vandvandrende insekter giver videnskabsfolk indsigt i, hvordan mikroplast skubbes under vandet
- Nye fodspor fra fusionsbegivenheden Gaia-pølse-enceladus
- Sådan finder du nuller af en funktion


