Forskere bruger maskinlæring til at søge i videnskabelige data
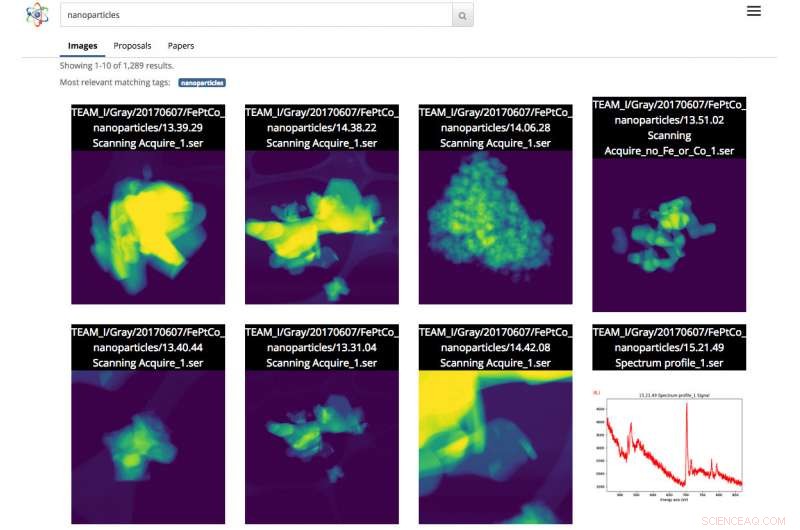
Skærmbillede af Science Search-grænsefladen. I dette tilfælde, brugeren foretog en billedsøgning af nanopartikler. Kredit:Gonzalo Rodrigo, Berkeley Lab
Efterhånden som videnskabelige datasæt øges i både størrelse og kompleksitet, evnen til at mærke, filtrer og søg denne syndflod af information er blevet en besværlig, tidskrævende og til tider umulig opgave, uden hjælp fra automatiserede værktøjer.
Med det i tankerne, et team af forskere fra Lawrence Berkeley National Laboratory (Berkeley Lab) og UC Berkeley udvikler innovative maskinlæringsværktøjer til at trække kontekstuel information fra videnskabelige datasæt og automatisk generere metadata-tags for hver fil. Forskere kan derefter søge i disse filer via en webbaseret søgemaskine efter videnskabelige data, kaldet Science Search, som Berkeley-holdet bygger.
Som et proof-of-concept, holdet arbejder med personale ved Department of Energy's (DOE) Molecular Foundry, placeret på Berkeley Lab, at demonstrere begreberne Science Search på billederne taget af anlæggets instrumenter. En betaversion af platformen er blevet gjort tilgængelig for Foundry-forskere.
"Et værktøj som Science Search har potentialet til at revolutionere vores forskning, " siger Colin Ophus, en Molecular Foundry-forsker i National Center for Electron Microscopy (NCEM) og Science Search Collaborator. "Vi er en skatteyder-finansieret national brugerfacilitet, og vi vil gerne gøre alle data bredt tilgængelige, snarere end det lille antal billeder, der er valgt til offentliggørelse. Imidlertid, i dag, de fleste af de data, der indsamles her, bliver kun rigtig set på af en håndfuld mennesker – dataproducenterne, inklusive PI (principal investigator), deres postdocs eller kandidatstuderende – for der er i øjeblikket ingen nem måde at gennemskue og dele dataene på. Ved at gøre disse rådata let søgbare og delbare, via internettet, Science Search kunne åbne dette reservoir af 'mørke data' for alle videnskabsmænd og maksimere vores anlægs videnskabelige effekt."
Udfordringerne ved at søge videnskabelige data
I dag, søgemaskiner bruges allestedsnærværende til at finde information på internettet, men søgning af videnskabelige data giver en anden række udfordringer. For eksempel, Googles algoritme er afhængig af mere end 200 spor for at opnå en effektiv søgning. Disse spor kan komme i form af nøgleord på en webside, metadata i billeder eller publikumsfeedback fra milliarder af mennesker, når de klikker på den information, de leder efter. I modsætning, videnskabelige data kommer i mange former, der er radikalt anderledes end en gennemsnitlig webside, kræver kontekst, der er specifik for videnskaben og mangler ofte også metadata til at give kontekst, der kræves for effektive søgninger.
På nationale brugerfaciliteter som Molecular Foundry, forskere fra hele verden søger om tid og rejser derefter gratis til Berkeley for at bruge ekstremt specialiserede instrumenter. Ophus bemærker, at de nuværende kameraer på mikroskoper på støberiet kan indsamle op til en terabyte data på under 10 minutter. Brugere skal derefter manuelt gennemsøge disse data for at finde kvalitetsbilleder med "god opløsning" og gemme disse oplysninger på et sikkert delt filsystem, som Dropbox, eller på en ekstern harddisk, som de til sidst tager med hjem for at analysere.
Ofte, de forskere, der kommer til Molecular Foundry, har kun et par dage til at indsamle deres data. Fordi det er meget trættende og tidskrævende manuelt at tilføje noter til terabyte af videnskabelige data, og der er ingen standard for at gøre det, de fleste forskere skriver blot stenografiske beskrivelser i filnavnet. Dette kan give mening for den person, der gemmer filen, men giver ofte ikke meget mening for andre.
"Manglen på rigtige metadata-etiketter forårsager i sidste ende problemer, når videnskabsmanden forsøger at finde dataene senere eller forsøger at dele dem med andre, " siger Lavanya Ramakrishnan, en ansat videnskabsmand i Berkeley Labs Computational Research Division (CRD) og co-principal investigator i Science Search-projektet. "Men med maskinlæringsteknikker, vi kan få computere til at hjælpe med, hvad der er besværligt for brugerne, herunder tilføjelse af tags til dataene. Så kan vi bruge disse tags til effektivt at søge i dataene."
For at løse metadataproblemet, Berkeley Lab-teamet bruger maskinlæringsteknikker til at udvinde det "videnskabelige økosystem" - inklusive instrumenttidsstempler, facilitets brugerlogfiler, videnskabelige forslag, publikationer og filsystemstrukturer - for kontekstuel information. Den samlede information fra disse kilder, inklusive tidsstemplet for eksperimentet, bemærkninger om den anvendte opløsning og filter og brugerens anmodning om tid, alle giver kritisk kontekstuel information. Berkeleys laboratorieteam har sammensat en innovativ softwarestak, der bruger maskinlæringsteknikker, herunder naturlig sprogbehandling, trækker kontekstuelle nøgleord om det videnskabelige eksperiment og automatisk skaber metadata-tags til dataene.
For proof-of-concept, Ophus delte data fra Molecular Foundry's TEAM 1 elektronmikroskop på NCEM, som for nylig blev indsamlet af facilitetspersonalet, med Science Search Team. Han meldte sig også frivilligt til at mærke et par tusinde billeder for at give maskinlæringsværktøjerne nogle etiketter, hvorfra han kunne begynde at lære. Selvom dette er en god start, Science Search co-principal investigator Gunther Weber bemærker, at de fleste succesrige maskinlæringsapplikationer typisk kræver betydeligt flere data og feedback for at levere bedre resultater. For eksempel, i tilfælde af søgemaskiner som Google, Weber bemærker, at træningsdatasæt oprettes og maskinlæringsteknikker valideres, når milliarder af mennesker verden over bekræfter deres identitet ved at klikke på alle billederne med gadeskilte eller butiksfacader efter at have indtastet deres adgangskoder, eller på Facebook, når de tagger deres venner i et billede.
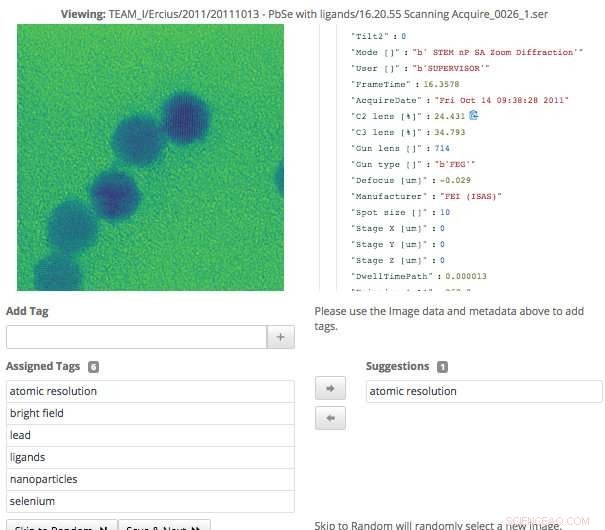
Dette skærmbillede af Science Search-grænsefladen viser, hvordan brugere nemt kan validere metadata-tags, der er blevet genereret via maskinlæring, eller tilføje oplysninger, der ikke allerede er blevet fanget. Kredit:Gonzalo Rodrigo, Berkeley Lab
"I tilfælde af videnskabelige data kan kun en håndfuld domæneeksperter oprette træningssæt og validere maskinlæringsteknikker, så et af de store vedvarende problemer, vi står over for, er et ekstremt lille antal træningssæt, " siger Weber, som også er stabsforsker i Berkeley Labs CRD.
For at overkomme denne udfordring, Berkeley Lab-forskerne brugte overførselslæring til at begrænse graderne af frihed, eller parametertællinger, på deres konvolutionelle neurale netværk (CNN'er). Transfer learning er en maskinlæringsmetode, hvor en model udviklet til en opgave genbruges som udgangspunkt for en model på en anden opgave. som giver brugeren mulighed for at få mere præcise resultater fra et mindre træningssæt. I tilfældet med TEAM I-mikroskopet, de producerede data indeholder information om, hvilken driftstilstand instrumentet var i på indsamlingstidspunktet. Med den information, Weber var i stand til at træne det neurale netværk på denne klassifikation, så det kunne generere denne funktionsmåde automatisk. Han frøs derefter det foldede lag af netværket, hvilket betød, at han kun skulle genoptræne de tæt forbundne lag. Denne tilgang reducerer effektivt antallet af parametre på CNN, giver holdet mulighed for at få nogle meningsfulde resultater fra deres begrænsede træningsdata.
Maskinlæring for at udvinde det videnskabelige økosystem
Ud over at generere metadata-tags gennem træningsdatasæt, Berkeley Lab-teamet udviklede også værktøjer, der bruger maskinlæringsteknikker til at udvinde det videnskabelige økosystem til datakontekst. For eksempel, dataindtagelsesmodulet kan se på et væld af informationskilder fra det videnskabelige økosystem – inklusive instrumenttidsstempler, brugerlogfiler, forslag og publikationer – og identificere fællestræk. Værktøjer udviklet på Berkeley Lab, der bruger naturlige sprogbehandlingsmetoder, kan derefter identificere og rangere ord, der giver kontekst til dataene og faciliterer meningsfulde resultater for brugerne senere. Brugeren vil se noget, der ligner resultatsiden for en internetsøgning, hvor indhold med mest tekst, der matcher brugerens søgeord, vises højere på siden. Systemet lærer også af brugerforespørgsler og de søgeresultater, de klikker på.
Fordi videnskabelige instrumenter genererer en stadigt voksende mængde data, alle aspekter af Berkeley-teamets videnskabelige søgemaskine skulle være skalerbare for at holde trit med hastigheden og omfanget af de datamængder, der produceres. Holdet opnåede dette ved at opsætte deres system i en Spin-instans på Cori-supercomputeren ved National Energy Research Scientific Computing Center (NERSC). Spin er en Docker-baseret edge-services-teknologi udviklet på NERSC, der kan få adgang til facilitetens højtydende computersystemer og lager på bagenden.
"En af grundene til, at det er muligt for os at bygge et værktøj som Science Search, er vores adgang til ressourcer på NERSC, " siger Gonzalo Rodrigo, en Berkeley Lab postdoc-forsker, der arbejder med de naturlige sprogbehandlings- og infrastrukturudfordringer i Science Search. "Vi skal opbevare, analysere og hente virkelig store datasæt, og det er nyttigt at have adgang til en supercomputing-facilitet til at udføre de tunge løft til disse opgaver. NERSC's Spin er en fantastisk platform til at køre vores søgemaskine, der er en brugervendt applikation, der kræver adgang til store datasæt og analytiske data, der kun kan gemmes på store supercomputing-lagringssystemer."
En grænseflade til validering og søgning af data
Da Berkeley Lab-teamet udviklede grænsefladen til, at brugere kunne interagere med deres system, de vidste, at det ville være nødt til at nå et par mål, herunder effektiv søgning og tillader menneskelig input til maskinlæringsmodellerne. Fordi systemet er afhængigt af domæneeksperter til at hjælpe med at generere træningsdata og validere maskinlæringsmodellens output, den nødvendige grænseflade for at lette det.
"Tag-grænsefladen, som vi udviklede, viser de originale data og metadata, der er tilgængelige, samt eventuelle maskingenererede tags vi har indtil videre. Ekspertbrugere kan derefter gennemse dataene og oprette nye tags og gennemgå alle maskingenererede tags for nøjagtighed, " siger Matt Henderson, som er Computer Systems Engineer i CRD og leder indsatsen for udvikling af brugergrænseflader.
For at lette en effektiv søgning efter brugere baseret på tilgængelig information, teamets søgegrænseflade giver en forespørgselsmekanisme for tilgængelige filer, forslag og papirer, som de Berkeley-udviklede maskinlæringsværktøjer har analyseret og udtrukket tags fra. Hvert anført søgeresultatelement repræsenterer en oversigt over disse data, med en mere detaljeret sekundær visning tilgængelig, herunder oplysninger om tags, der matchede denne vare. Holdet undersøger i øjeblikket, hvordan man bedst kan inkorporere brugerfeedback for at forbedre modellerne og tags.
"At have evnen til at udforske datasæt er vigtigt for videnskabelige gennembrud, og det er første gang, at noget lignende Science Search er blevet forsøgt, " siger Ramakrishnan. "Vores ultimative vision er at bygge fundamentet, der i sidste ende vil understøtte en 'Google' for videnskabelige data, hvor forskere endda kan søge i distribuerede datasæt. Vores nuværende arbejde giver det nødvendige grundlag for at nå den ambitiøse vision."
"Berkeley Lab er virkelig et ideelt sted at bygge et værktøj som Science Search, fordi vi har en række brugerfaciliteter, ligesom Molecular Foundry, der har årtiers data, der ville give endnu mere værdi for det videnskabelige samfund, hvis dataene kunne søges og deles, " tilføjer Katie Antipas, som er hovedefterforsker af Science Search og leder af NERSCs dataafdeling. "Derudover har vi stor adgang til maskinlæringsekspertise inden for Berkeley Lab Computing Sciences-området samt HPC-ressourcer hos NERSC for at opbygge disse muligheder."
 Varme artikler
Varme artikler
-
 Tyskland godkender gruppesøgsmål inden diesel-deadlineÆndringen vil gøre det lettere for tyske VW-ejere at søge erstatning Den tyske regering godkendte onsdag et lovudkast, der tillader gruppesøgsmål i amerikansk stil, åbner døren for, at chauffører
Tyskland godkender gruppesøgsmål inden diesel-deadlineÆndringen vil gøre det lettere for tyske VW-ejere at søge erstatning Den tyske regering godkendte onsdag et lovudkast, der tillader gruppesøgsmål i amerikansk stil, åbner døren for, at chauffører -
 Ransomware-helten erkender sig skyldig i amerikanske anklager om hackingMarcus Hutchins (R), set forud for en retsoptræden i 2017, blev hyldet som en helt for at dæmme op for WannaCry ransomware-udbruddet, men senere sigtet i USA for at skabe malware, der kunne angribe ba
Ransomware-helten erkender sig skyldig i amerikanske anklager om hackingMarcus Hutchins (R), set forud for en retsoptræden i 2017, blev hyldet som en helt for at dæmme op for WannaCry ransomware-udbruddet, men senere sigtet i USA for at skabe malware, der kunne angribe ba -
 Lav værktøjerne til at forbinde isiXhosa og isiZulu med den digitale tidsalderSoftwareværktøjer kan tage flere sprog til helt nye rum. Kredit:Zubada/Shutterstock Vi lever i en verden, hvor der tales omkring 7000 sprog, og en, hvor informations- og kommunikationsteknologier
Lav værktøjerne til at forbinde isiXhosa og isiZulu med den digitale tidsalderSoftwareværktøjer kan tage flere sprog til helt nye rum. Kredit:Zubada/Shutterstock Vi lever i en verden, hvor der tales omkring 7000 sprog, og en, hvor informations- og kommunikationsteknologier -
 Coronavirus kan bremse Apple iPhone-forsendelserKredit:CC0 Public Domain Coronavirussen kan påvirke de teknologiske produkter, der dukker op på dit dørtrin. De fleste af de populære bedst sælgende produkter, ligesom Apple iPhone, iPad og Amazo
Coronavirus kan bremse Apple iPhone-forsendelserKredit:CC0 Public Domain Coronavirussen kan påvirke de teknologiske produkter, der dukker op på dit dørtrin. De fleste af de populære bedst sælgende produkter, ligesom Apple iPhone, iPad og Amazo
- Palæontologer identificerer nye arter af mosasaurer
- Luftfarts bidrag til at reducere klimaændringer vil sandsynligvis være lille
- Brandslukningsskum efterlader giftig arv i drikkevand nær militærbaser
- Hvordan finder en Hummingbird Food?
- Tyfusforgiftning har en sød tand
- Peberplukkerobot demonstrerer sine færdigheder inden for automatisering af drivhusarbejder


