Forskere foreslår effektivitetsregler for at forbedre brugen af ny genredigeringsteknologi
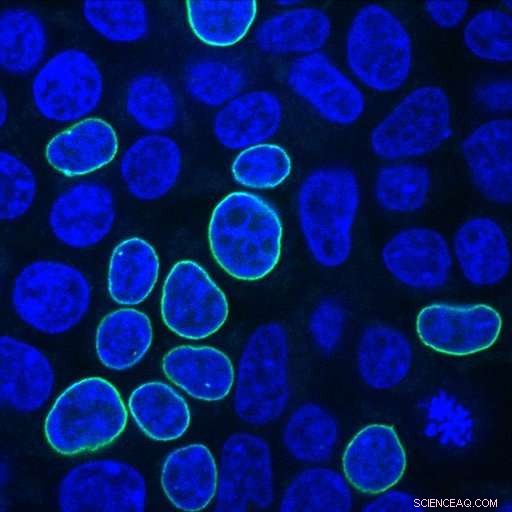
Humane embryonale nyreceller lyser grønt efter reparation af et CRISPR-induceret DNA-brud med et PCR-fragment, der koder for et fluorescerende protein og homologiarme med 33 nukleotider. Kredit:Alexandre Paix
Johns Hopkins-forskere har udviklet en strømlinet metode og medfølgende effektivitets-"regler" for at introducere nye DNA-sekvenser i celler efter brug af genskæringsværktøjet kendt som CRISPR. Forskerne siger, at metoden, som de baserede på test med museembryoner og tusindvis af menneskelige celler, kunne forbedre konsekvens og effektivitet af genomredigering.
Den nye metode og dens udvikling er beskrevet online i den 28. november i Proceedings of the National Academy of Sciences .
"CRISPR er et værktøj til at hjælpe forskere med at ændre genomet, forudsige resultatet af visse egenskaber og studere dem, men selve værktøjet skaber kun brud i genomet. Det styrer ikke, hvordan en ny DNA-sekvens indsættes i genomet, " siger Geraldine Seydoux, Ph.D., Huntington Sheldon-professor i medicinsk opdagelse ved Institut for Molekylærbiologi og Genetik og vicedekan for grundforskning ved Johns Hopkins University School of Medicine, og en efterforsker ved Howard Hughes Medical Institute.
"Vi satte os for at undersøge, hvordan celler reparerer brud fremkaldt af CRISPR med det mål at bruge cellens naturlige DNA-reparationsproces til at introducere nye sekvenser i genomet. Vi var overraskede over at opdage, at celler let vil kopiere sekvenser fra fremmed DNA for at reparere DNA-brud. , så længe de fremmede DNA'er er lineære, " Seydoux tilføjer. "Ved at studere, hvordan fremmede DNA-fragmenter kopieres under reparationsprocessen, vi fandt på nogle enkle regler for at gøre genomredigering så effektiv som muligt, optimere værktøjet, og gør det med tillid."
CRISPR, som står for clustered regularly interspaced short palindromic repeat, har vundet popularitet blandt videnskabsmænd i de sidste fem år som et værktøj til effektivt at skære DNA. Det blev tilpasset til brug i pattedyrceller fra en naturlig viral forsvarsproces i bakterieceller, der involverer at skabe dødelige snit i viralt DNA. I det væsentlige, værktøjet er et strømlinet sæt molekylære "saks".
Den fremherskende tro, blandt videnskabsmænd, er, at celler reparerer DNA-brud ved at indsætte et tilfældigt sæt nukleotider, de kemiske byggesten i DNA. Dette ødelægger normalt ethvert gen, der er placeret på det sted, hvor DNA'et er brudt.
Det er også velkendt for videnskabsmænd, at lejlighedsvis, celler bruger en anden kilde - en sekvens fra et andet stykke DNA, eller "donor"-DNA - for at forsegle bruddet i DNA. Imidlertid, den nye "donor"-sekvens kan ikke indsættes af sig selv i et tomt rum i genomet.
I stedet, det nye donor-DNA har brug for en slags tape i hver ende for at hjælpe det med at holde fast i det mellemrum, der er lavet af snittet. Forskere omtaler dette bånd som donor-DNA's "homologi"-arme.
Homologiarmene består af nukleotider, der overlapper de intakte dele af DNA'et med matchende genetisk kode. Dette hjælper donor-DNA'et til at "klistre" til det intakte DNA.
Endnu, videnskabsmænd betragtede brugen af donor-DNA som en ineffektiv måde at reparere genomet på, forudsat at det krævede lange homologiarme, især når man indsætter en lang DNA-sekvens, og enkeltstrenget eller cirkulært DNA, som er svære at tilberede i lange størrelser.
Efterhånden som forskerne fik mere erfaring med CRISPR, Seydoux siger, "Der opstod spørgsmål om de optimale designregler for donor-DNA og længden af homologiarmene."
Søger svar på disse spørgsmål, Johns Hopkins-forskerne indsatte forskellige kombinationer af donor-DNA i humane embryonale nyreceller, kendt for deres evne til at vokse godt og for deres hyppige brug i kræftforskning. Forskerne brugte donor-DNA med et gen, der koder for et fluorescerende protein, som lyser grønt i cellens kernemembran, når genindsættelsen lykkes.
Johns Hopkins forskningsmedarbejder Alexandre Paix fandt ud af, at lineære DNA-fragmenter fungerer meget godt som donorer, og er to til fem gange mere effektive end cirkulære DNA'er (kendt som plasmider) i humane celler. "Lineær DNA er meget let at forberede i laboratoriet, ved hjælp af PCR, " siger Paix, med henvisning til polymerasekædereaktionsværktøjer, som bruges til at amplificere DNA.
Paix testede også forskellige længder af homologiarme. Han fandt ud af, at det søde sted for homologiarme er omkring 35 nukleotider i længden, meget kortere end forskerne typisk bruger.
Specifikt, det blev fundet, at homologiarme med en længde på 33 til 38 nukleotider var lige så vellykkede som dem med 518 nukleotider, giver mellem 10 og 20 procent vellykkede redigeringer under optimale forhold. I modsætning, da forskerne testede homologiarme på 15 og 16 nukleotider lange, succesraterne for indsættelse faldt med det halve. De gentog disse resultater på tre forskellige steder i det menneskelige genom.
De fandt også ud af, at den nyligt indsatte sekvens, uden at tælle homologi-armene, kan være op til 1, 000 nukleotider i længden.
Holdet opnåede succesrater mellem 10 og 50 procent med inserts varierende fra 57 til 993 nukleotider i længden. Kortere sekvenser blev mere vellykket indsat end længere. For eksempel, nye sekvenser, der var 57, 714 og 993 nukleotider lange blev med succes indsat 45,4, 23,5 og 17,9 procent af tiden, henholdsvis. Ud over 1, 000 nukleotider, nye indsatser med 1, 122 og 2, 229 nukleotider havde ringe succes - omkring 0,5 procent af tiden. "I den størrelse det bliver meget vanskeligt at introducere den mængde donor-DNA, der er nødvendig for redigering. Celler har en tendens til at 'kvæle' i så meget DNA, " siger Seydoux.
Endelig, holdet fandt også, at succesraten for redigering topper, når den nye sekvens er placeret inden for 30 nukleotider fra CRISPR cut-stedet. "Ud over 30 nukleotider, indsættelsen kan ikke fungere, " siger Seydoux.
"Disse parametre bør rumme de fleste gener, som videnskabsmænd søger at redigere. Faktisk, de fleste eksperimenter involverer kun redigering af to til tre nukleotider tæt på CRISPR-udskæringsstedet, " tilføjer Seydoux.
Forskerholdet testede også, om den samme tilgang kunne fungere i museembryoner. Ved at bruge et PCR-fragment med 36-nukleotid homologi arme, holdet indsatte med succes en 739 nukleotider lang sekvens, der koder for et fluorescerende protein, i 27 af 87 (31 procent) museembryoner.
Seydoux's forskerhold bruger allerede reparationsreglerne til at studere DNA i Caenorhabditis elegans, en art af orme, og forskerne undersøger, om reparationsreglerne gælder for andre typer menneskeceller.
Før retningslinjerne bliver bredt vedtaget, Seydoux siger, at de bør testes i flere menneskelige celletyper og andre organismer.
 Varme artikler
Varme artikler
-
 Dino-dræbende asteroide fremskyndede fugleudviklingenEn gennemsnitlig fugl fra umiddelbart før K-Pg-grænsen vejede omkring 1 kg, ligner en Gulnæbbet And (til venstre); en gennemsnitlig fugl vejer i dag omkring 37 g, størrelsen af Cape Weaver (til højr
Dino-dræbende asteroide fremskyndede fugleudviklingenEn gennemsnitlig fugl fra umiddelbart før K-Pg-grænsen vejede omkring 1 kg, ligner en Gulnæbbet And (til venstre); en gennemsnitlig fugl vejer i dag omkring 37 g, størrelsen af Cape Weaver (til højr -
 Hvad er der i din hvede? Forskere sammensætter genomet af den mest almindelige brødhvedeKredit:CC0 Public Domain Johns Hopkins-forskere rapporterer, at de med succes har brugt to separate genteknologier til at samle den mest komplette genomsekvens til dato af Triticum aestivum, den m
Hvad er der i din hvede? Forskere sammensætter genomet af den mest almindelige brødhvedeKredit:CC0 Public Domain Johns Hopkins-forskere rapporterer, at de med succes har brugt to separate genteknologier til at samle den mest komplette genomsekvens til dato af Triticum aestivum, den m -
 Er der en sammenhæng mellem træning og lykke?Hvis du ikke har et stort grin i ansigtet fra tanken om at løbe på et løbebånd, måske har du aldrig nået det endorfin højt. Se flere sundhedsbilleder. © iStockphoto.com/LajosRepasi Hvis du nogensinde
Er der en sammenhæng mellem træning og lykke?Hvis du ikke har et stort grin i ansigtet fra tanken om at løbe på et løbebånd, måske har du aldrig nået det endorfin højt. Se flere sundhedsbilleder. © iStockphoto.com/LajosRepasi Hvis du nogensinde -
 Forskeren studerer fugle, der bryder alle parringsreglerPeter Dunn, UWM fremragende professor i biologiske videnskaber, har studeret parringsvaner for træspurve og andre fugle på UWM Field Station i Cedarburg Bog i mere end 20 år. Værket har givet nogle ov
Forskeren studerer fugle, der bryder alle parringsreglerPeter Dunn, UWM fremragende professor i biologiske videnskaber, har studeret parringsvaner for træspurve og andre fugle på UWM Field Station i Cedarburg Bog i mere end 20 år. Værket har givet nogle ov
- Hjælper eller skader genoprettende retfærdighed ofre?
- Lille højspændings transmissionselektronmikroskop bygget i Japan
- Forskere opnår den ultimative opløsningsgrænse i fluorescensmikroskopi
- Ødelægger havstigninger husværdier ved kysten? Svaret:Måske
- Kinesiske aktivister protesterer mod brugen af traditionelle behandlinger - de vil have lægeviden…
- Klimapolitikker alene vil ikke redde Jordens mest forskelligartede tropiske skove


